本帖最后由 薛定谔了么 于 2025-7-3 11:29 编辑
蛋白质是许多生物活动的主要 “执行者”,人类疾病疗法的开发,大多围绕对蛋白质功能的理解与调控展开 。像布洛芬这类小分子药物,通常通过抑制或促进蛋白质功能,或调节其与其他生物分子的相互作用来发挥作用。长期以来,开发有效的 “虚拟筛选” 方法(即通过计算而非实验室实验来评估潜在小分子)一直是研究热点。然而,该问题长期存在一个挑战:如何构建一种在实际应用中表现良好的方法。
该本研究结合 DNA 编码小分子库的物理筛选与图卷积神经网络(GCNN)的虚拟筛选,成功发现了具有生物活性的分子。这项研究促成了 “化学组计划” 的诞生 —— 这是谷歌加速科学团队与 ZebiAI 的合作项目,将助力发现更多用于生物学研究的小分子化学探针。2020年6月11日,该团队与 X-Chem Pharmaceuticals 合作在《药物化学杂志》发表了《DNA 编码库的机器学习:命中发现的新范式》,文中展示了一种高效发现化学探针的新方法。
理解支持生命和引发疾病的生物网络是一项极其复杂的任务。研究这些过程的一种方法是使用化学探针 —— 这类小分子不一定能直接作为药物,但能选择性抑制或促进特定蛋白质的功能。当研究某一生物系统(如培养皿中的癌细胞)时,可在特定时间加入化学探针,通过观察目标蛋白质活性增强或减弱时生物系统的不同反应来获取信息。然而,尽管化学探针在基础生物医学研究中作用显著,但目前已知的人类蛋白质中,仅有 4% 拥有对应的化学探针。
寻找化学探针的过程与小分子药物发现的早期阶段类似:针对目标蛋白质,扫描小分子空间以找到可进一步测试的 “命中” 分子。机器人辅助的高通量筛选(可对数十万甚至数百万分子进行物理测试)是现代药物研究的基石。然而,可轻松购买的小分子数量(约 1.2×10⁹个)远多于高通量筛选的处理能力,而潜在可合成的小分子数量(估计在 10²⁰到 10⁶⁰之间)更是庞大。“虚拟筛选” 有望快速高效地探索这一广阔的分子空间,大幅加速治疗化合物的发现。
筛选过程的物理部分使用 DNA 编码小分子库(DELs),其中包含多种不同的小分子,每个分子都连接着一段 DNA 片段,作为该分子的唯一条形码。尽管这项基础技术已存在数十年,但库的质量和筛选过程是产生有意义结果的关键。
DELs 的设计巧妙解决了生化领域的一个挑战:如何高效收集大量分子。其构建过程如下:首先,将 DNA “条形码”(如图中带数字的螺旋结构所示)连接到带有共同化学手柄(如 NH₂)的小分子片段(蓝色形状)上;同时,将另一组带有不同化学手柄(如 OH)的小分子片段(橙色形状)与 DNA 片段混合。随后,通过两步反应,两组化学片段在共同的化学手柄处反应融合,DNA 片段也随之连接,形成每个分子独有的连续条形码。最终,通过 2N 次操作可生成 N² 个独特分子,每个分子都由其唯一的 DNA 条形码标识。使用更多片段或更多循环,可轻松构建包含数百万甚至数十亿个不同分子的库。
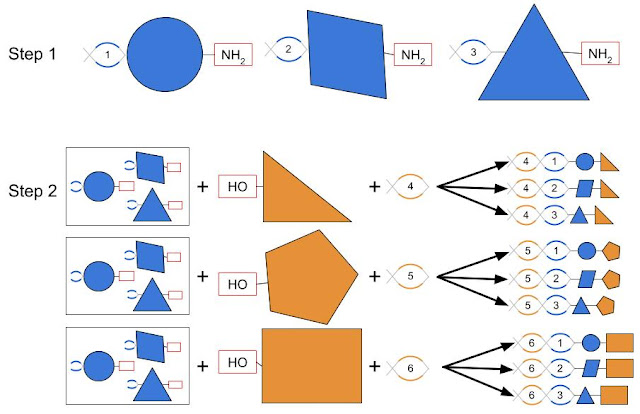
创建 DNA 编码小分子库过程
生成库后,可通过将 DEL 与目标蛋白质混合,并洗去未结合的小分子,来寻找能与该蛋白质结合的小分子。对剩余 DNA 条形码进行测序,可获得数百万条 DNA 片段的单独读数,经仔细处理后,可估算原始 DEL 中数十亿分子里哪些与蛋白质发生了相互作用。
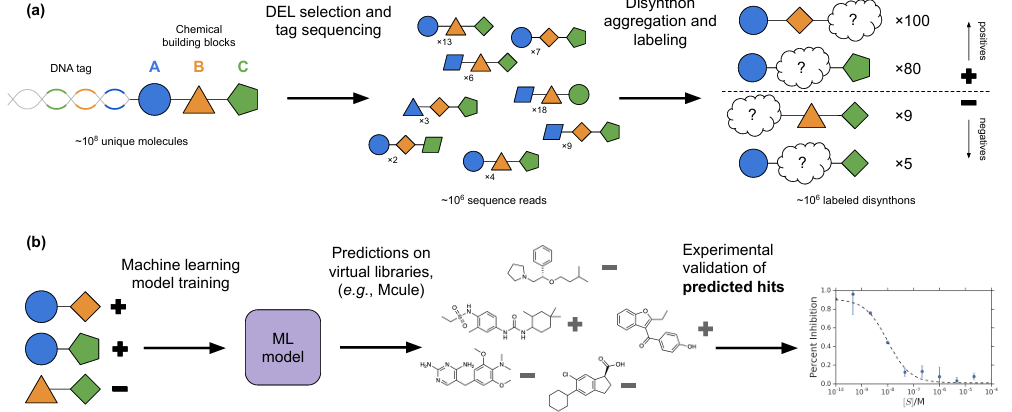
基于 DNA 编码库(DEL)数据训练机器学习模型的流程示意图
物理筛选为机器学习分类器提供了正负样本,用于训练模型预测任意选择的小分子是否会与目标蛋白质结合。简化来看,筛选结束后剩余的小分子为正样本,其余为负样本。研究使用了图卷积神经网络(GCNN)—— 一种专为小分子等类图输入设计的神经网络。
本研究使用 DEL 训练的模型,对三种不同的蛋白质进行了物理筛选:sEH(一种水解酶)、ERα(一种核受体)和 c-KIT(一种激酶),并利用 GCNN 模型对 Mcule 和 X-Chem 的可按需合成大型分子库进行虚拟筛选,以识别预测与各目标具有亲和力的多样化分子。研究团队将 GCNN 模型的结果与随机森林(RF)模型(一种使用标准化学指纹的常见虚拟筛选方法,作为基线)进行了比较,发现 GCNN 模型在发现更有效候选分子方面显著优于 RF 模型。
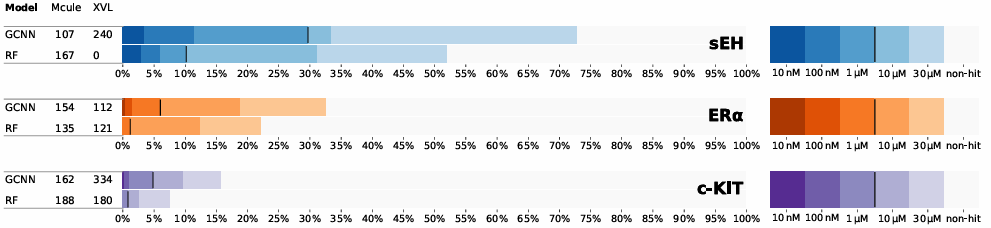
三种不同的蛋白质靶点比较两种不同机器学习模型(图卷积神经网络 GCNN 和随机森林 RF )的预测结果
值得注意的是,与许多其他虚拟筛选应用不同,该模型结果可直接自动化或轻松实现自动化来选择待测试分子,无需训练有素的化学家对最有希望的分子进行审查和选择。此外,该研究在三个目标上测试了近 2000 个分子,这是当时已发表的最大规模的虚拟筛选前瞻性研究。这不仅对上述命中率提供了高置信度,还允许仔细检查命中分子的多样性以及模型对不同分子的实用性。
文章改编转载自:谷歌研究院(Google Research)官方博客
原文链接:https://research.google/blog/unlocking-the-chemome-with-dna-encoded-chemistry-and-machine-learning/
论文链接:Machine Learning on DNA-Encoded Libraries: A New Paradigm for Hit Finding | Journal of Medicinal Chemistry | 






















