本帖最后由 Jack小新 于 2025-7-24 17:43 编辑
本研究利用变分自编码器(VAE)设计人工线粒体靶向序列(AMTS)。经训练生成的序列中 90.14% 具线粒体靶向功能,70.4% 含 TOM20 识别基序。在四种真核生物中验证 41 条新肽段,成功率 50%-100%,HEK293 细胞中肽段可被剪切。Dual-VAE 生成 62 条双靶向序列,提示其可能源于 MTS 进化。应用中,线粒体定位 β- 丙氨酸途径酶使 3 - 羟基丙酸产量提升 1.62 倍,嵌合 MTS 使 HEM1 靶向效率提升 4.76 倍。
正如人体每个器官各司其职,细胞内部的「细胞器」也是协同维持细胞的整体运作。这些膜包被的细胞器为大量核编码蛋白提供了最适理化环境,使其能够执行关键生物学功能。
为维持细胞组织与进程,细胞内部存在确保蛋白质准确送达的复杂机制 —— 蛋白质通过独特的氨基酸靶向序列被标记并运往特定细胞器。
线粒体作为细胞的 “能量工厂”,在能量代谢、物质合成及细胞凋亡中扮演核心角色,其功能异常与代谢疾病、线粒体遗传病等密切相关。精准靶向线粒体递送蛋白质或药物,是代谢工程优化和疾病治疗的关键手段。然而,天然线粒体靶向序列(MTS)的稀缺性和局限性(如靶向效率依赖载体蛋白、易饱和导入机制等),严重制约了相关研究与应用。
近期,伊利诺伊大学厄巴纳 - 香槟分校的研究团队在《Nature Communications》发表的成果,通过生成式人工智能技术突破了这一瓶颈,为线粒体靶向序列的设计与应用开辟了新路径。

一、MTS 的固有挑战与生成式 AI 的解决方案
天然 MTS 是核编码线粒体蛋白 N 端的短肽段,通过 TOM/TIM 复合体介导的识别与转运,引导蛋白进入线粒体基质,随后被线粒体加工肽酶(MPP)切割,使蛋白正确折叠。其功能依赖三大关键特征:正电荷特性、两亲性(兼具亲水与疏水区域)及易形成 α 螺旋结构。但目前已鉴定的 MTS 数量极少,重复使用会导致导入机制饱和、与内源蛋白竞争,甚至引发遗传不稳定性(如同源重组)。
为解决这一问题,研究团队采用变分自编码器(VAE)—— 一种能从数据中学习潜在模式的深度生成模型。VAE 通过编码器将输入的 MTS 序列转化为潜在空间向量,再通过解码器重构序列,最终可从潜在空间采样生成全新序列。训练数据涵盖 56,660 条 MTS(来自 Swiss-Prot 数据库及 TargetP 2.0 预测结果),经筛选去重后,确保序列长度在 11-69 个氨基酸,覆盖真核生物多样性。
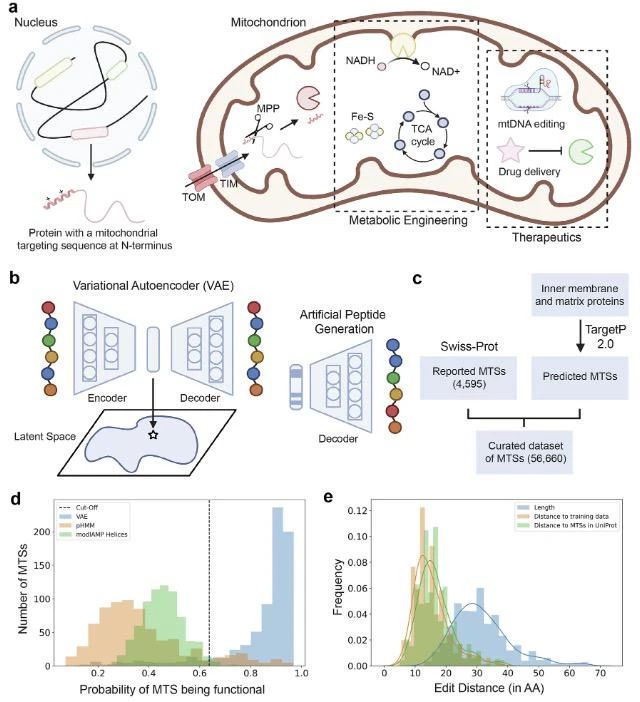
图示:用于生成线粒体靶向序列 (MTS) 的变分自编码器。
二、VAE 生成 MTS 的功能验证
VAE 生成的人工线粒体靶向序列(AMTS)经严格验证,展现出优异的功能性与多样性:
计算验证:通过 DeepLoc 2.0 预测,90.14% 的 VAE 生成序列可有效靶向线粒体,远高于传统方法(如 profile Hidden Markov Model 的 12.2%、modlAMP 的 1.37%)。序列分析显示,70.4% 的 AMTS 含 TOM20 识别基序(φχβφφ,φ 为疏水残基,β 为碱性残基),提示其通过经典 TOM/TIM 途径导入。
序列多样性:生成序列与天然 MTS 的编辑距离(氨基酸突变数)达 10-15,避免了同源重组风险,且 UMAP 分析显示其覆盖天然 MTS 的全部序列空间,填补了未探索的功能区域。
体内验证:研究设计了基于 UniRep 嵌入和 k 近邻的采样策略,优先筛选适配特定生物的 AMTS。在酿酒酵母(S. cerevisiae)、人 HEK293 细胞、本氏烟草(N. benthamiana)和红冬孢酵母(R. toruloides)中,41 条新肽段的靶向成功率达 50%-100%。例如,HEK293 细胞中 8 条 AMTS 均能与线粒体染料共定位,且 Western blot 证实其在体内被成功切割。
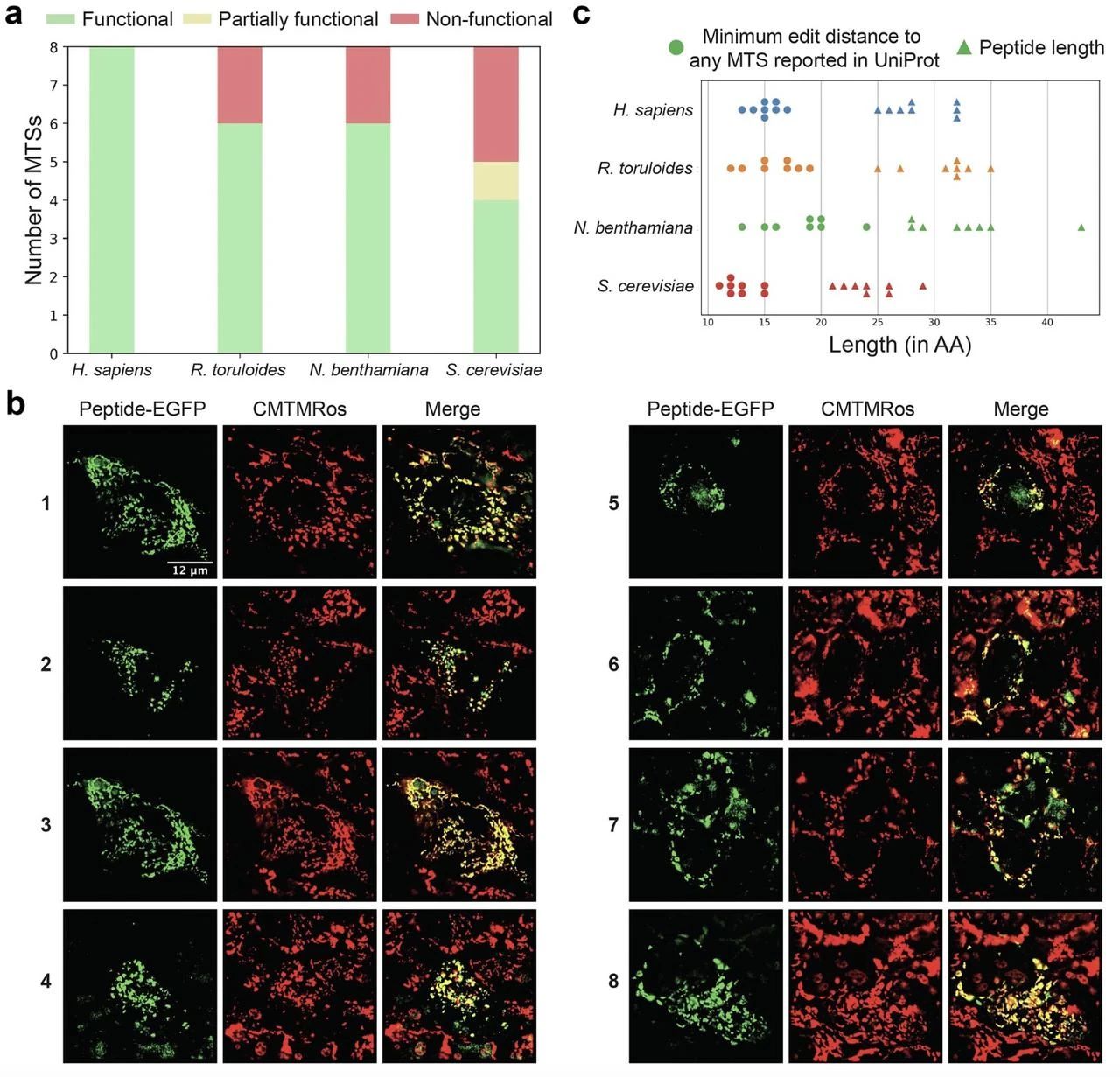
图示:四种真核生物中人工线粒体靶向序列(AMTS)的特征分析。
三、双靶向序列的设计与进化启示
部分生成序列显示出同时靶向线粒体和叶绿体的潜力。研究团队基于植物界的 MTS 和叶绿体靶向序列(CTS),训练了 Dual-VAE 模型,通过潜在空间线性插值生成 62 条双靶向序列。特征分析揭示了从 MTS 到 CTS 的平滑过渡:
氨基酸组成:丝氨酸(S)和亮氨酸(L)增加,精氨酸(R)减少(精氨酸是叶绿体靶向特异性的关键抑制因子);
结构变化:α 螺旋占比下降,线圈结构(无规则卷曲)增加;
长度差异:CTS 更长,提示可能通过插入片段扩展功能。
基于这些变化,研究提出假说:双靶向序列更可能从 MTS 进化而来 —— 通过积累突变调整电荷与结构,而从 CTS 进化则需额外插入片段以适配线粒体加工机制。这为理解植物细胞器靶向的进化提供了新视角。
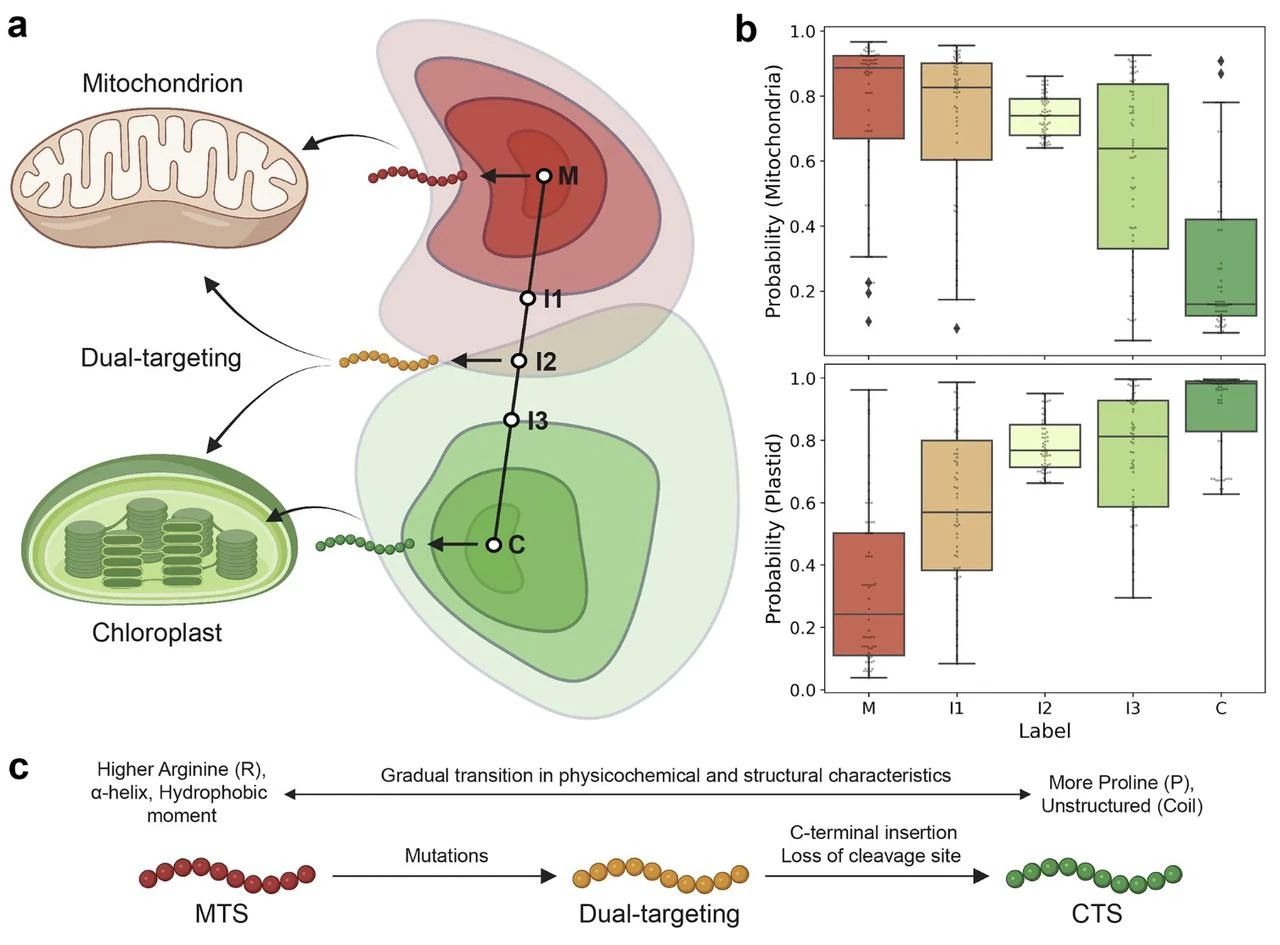
图示:利用 Dual-VAE 设计双靶向肽。
四、应用场景:代谢工程与蛋白递送的突破
研究通过两个案例验证了 AMTS 的实用价值:
3 - 羟基丙酸(3-HP)产量提升:将 β- 丙氨酸途径的 3 个关键酶PAND、BAPAT和YDFG,通过 AMTS 定位到线粒体,利用线粒体中丰富的前体物质(天冬氨酸),使 3-HP 产量达 2.76 g/L,较胞质途径(1.70 g/L)提升 1.62 倍。
HEM1 靶向效率优化:5 - 氨基乙酰丙酸合酶(HEM1)是血红素合成的限速酶,其靶向缺陷与人类血液病相关。通过嵌合 AMTS(如 AMTS3、AMTS131 与 COX4 组合),靶向效率最高提升 4.76 倍,且序列顺序和数量显著影响效果。
五、总结与展望
该研究证明,VAE 等生成式 AI 模型能高效设计多样化、功能性的 MTS 及双靶向序列,不仅为线粒体生物学基础研究提供了工具,更推动了代谢工程和疾病治疗的应用。未来,结合条件 VAE、蛋白质语言模型及高通量验证技术,有望进一步提升序列设计的精准性与应用范围,为细胞器靶向研究注入新动能。
文章改编转载自微信公众号:ScienceAI
原文链接:https://mp.weixin.qq.com/s/w4itwZFtVhVuvQKLIWuM3Q
相关报道:https://phys.org/news/2025-05-harnessing-generative-ai-mitochondrial-toolkit.html | 






















