本帖最后由 Akkio 于 2025-11-4 23:31 编辑
表于 Nature Biomedical Engineering 的《 High‑throughput evaluation of in vitro CRISPR activities enables optimized large‑scale multiplex enrichment of rare variants 》开发了两种体外高通量测定方法 Cut‑seq1 和 Cut‑seq2 ,用于评估成千上万对 sgRNA–靶序列配对的 Cas9 切割效率。结果显示,体外切割指数与细胞内 indel 频率相关性仅约 r≈0.27,但 PAM 兼容性的相关性接近 r≈0.99。基于约 18 万组数据构建的深度学习模型预测切割比值的相关性达 r≈0.90。利用该模型选择优化 sgRNA,并通过 CLOVE‑seq 实现平均约 900 倍的稀有变体等位基因频率富集,超过 80% 的突变被检测到。
以往针对大量靶序列与导向RNA(guide RNA, gRNA)的高通量CRISPR活性评估,多依赖插入–缺失突变(indel)频率而非切割效率。研究人员开发了两种高通量体外测定方法——Cut-seq1 与 Cut-seq2,可评估成千上万甚至上十万种Cas9-gRNA–靶序列配对的切割效率。结果显示,体外切割效率与细胞中indel频率相关性较低,但与PAM序列兼容性高度一致。
基于这些大规模体外切割效率数据,研究人员构建了DeepCut深度学习模型,用于识别最优单导向RNA(sgRNA),可在噪声序列存在时仍特异切割目标序列。进一步,研究人员开发了CLOVE-seq(Cleavage for Large-scale Optimized Variant Enrichment Sequencing)方法,通过Cas9介导的特异性切割实现稀有变体的多重富集。该体系不仅提升了对CRISPR核酸酶活性的理解,也为多种生物医学场景下稀有变体检测提供了可扩展的技术路线。
在哺乳动物细胞中进行的高通量 sgRNA–靶序列活性评估使得机器学习模型能准确预测 CRISPR 活性。然而,这些评估通常依赖于 indel 频率,而非直接的切割效率。细胞内的 indel 频率受到非同源末端连接误差率、染色质状态及 DNA 可及性等多重因素影响。因此,在可控条件下直接测量切割效率(如体外实验)对于理解 CRISPR 生物学至关重要。但此前的体外评估方法通常仅能分析少数 sgRNA,难以实现大规模并行评估。
为此,研究人员设计了可容纳数十万 sgRNA–靶序列对的体外体系,并进一步结合深度学习模型,实现对 sgRNA 切割活性、选择性及稀有变体富集的全面分析。
|| 方法概述
研究人员利用固定化的大肠杆菌作为反应单元,构建了含有 2,000 至 120,000 对 sgRNA–靶序列的质粒文库(T7_2k 与 T7_120k)。通过体外转录与 Cas9 反应,结合接头连接与深度测序,研究人员建立了 Cut-seq1 方法以量化 Cas9 切割指数。进一步引入识别位点酶 EcoRV,使得未被切割的片段也能被检测,从而形成改进版本 Cut-seq2,以切割比值指数更准确反映酶活性。
此外,研究人员设计了针对癌症相关突变的噪声序列(WT)与稀有变体(MT)文库,用以评估不同 sgRNA 的选择性和富集效率。
|| 结果
Cut-seq1 的开发与验证
研究人员首先利用固定化细菌实现体外 CRISPR–Cas9 反应的空间分隔,并构建了 T7 启动子驱动的配对文库。深度测序显示两次生物学重复间的切割指数相关性极高(r = 0.95)。通过测试不同菌株与固定方法,确定了缺乏核酸内切酶 A 的大肠杆菌(EC100)和低温甲醇固定为最优条件。实验验证表明,反应体系中切割反应在单个细胞中实现良好分隔。

图1 Cut-seq1的开发与验证
体外与细胞内切割活性对比
为了比较体外切割效率与细胞内 indel 频率的关系,研究人员构建了使用 U6 启动子的文库(U6_120k),并在多种细胞系(HEK293T、HeLa、Hepa 1–6、B16-F10)中测定。结果显示不同细胞间的 indel 频率高度相关,但体外切割指数与细胞内 indel 频率的相关性较低(r ≈ 0.27),而 PAM 相容性在体外与细胞中则几乎一致(r ≈ 0.99)。这表明 indel 频率受细胞环境影响较大,而 PAM 识别规律具有普适性。
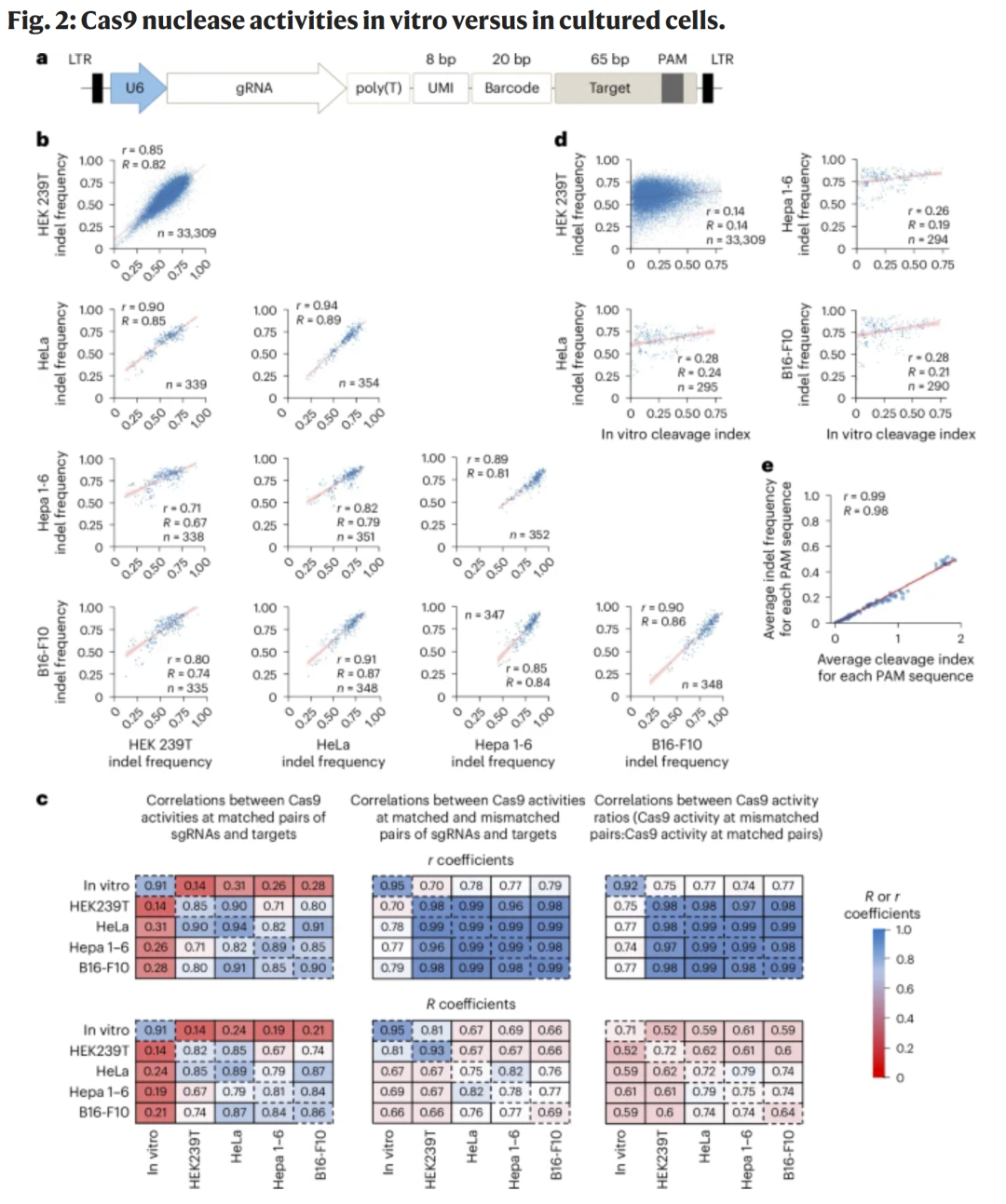
图2 Cas9在体外与细胞内的活性比较
sgRNA 差异性选择性与 Cut-seq2 的建立
研究人员构建了包含 30,772 对 sgRNA–靶序列的 Cut1_30k 文库,用以评估不同 Cas9 变体(SpCas9、HF1、SuperFi、evoCas9)及其在 1-bp 差异序列下的切割选择性。结果显示,高保真变体 HF1 与 SuperFi 具有更优的选择性指数(SI),且多数最佳区分 sgRNA 并非完全匹配,而是带有单碱基错配的“差异型 sgRNA”。基于此,研究人员改进为 Cut-seq2,可同时检测被切割与未切割序列,以切割比值指数更准确反映活性。
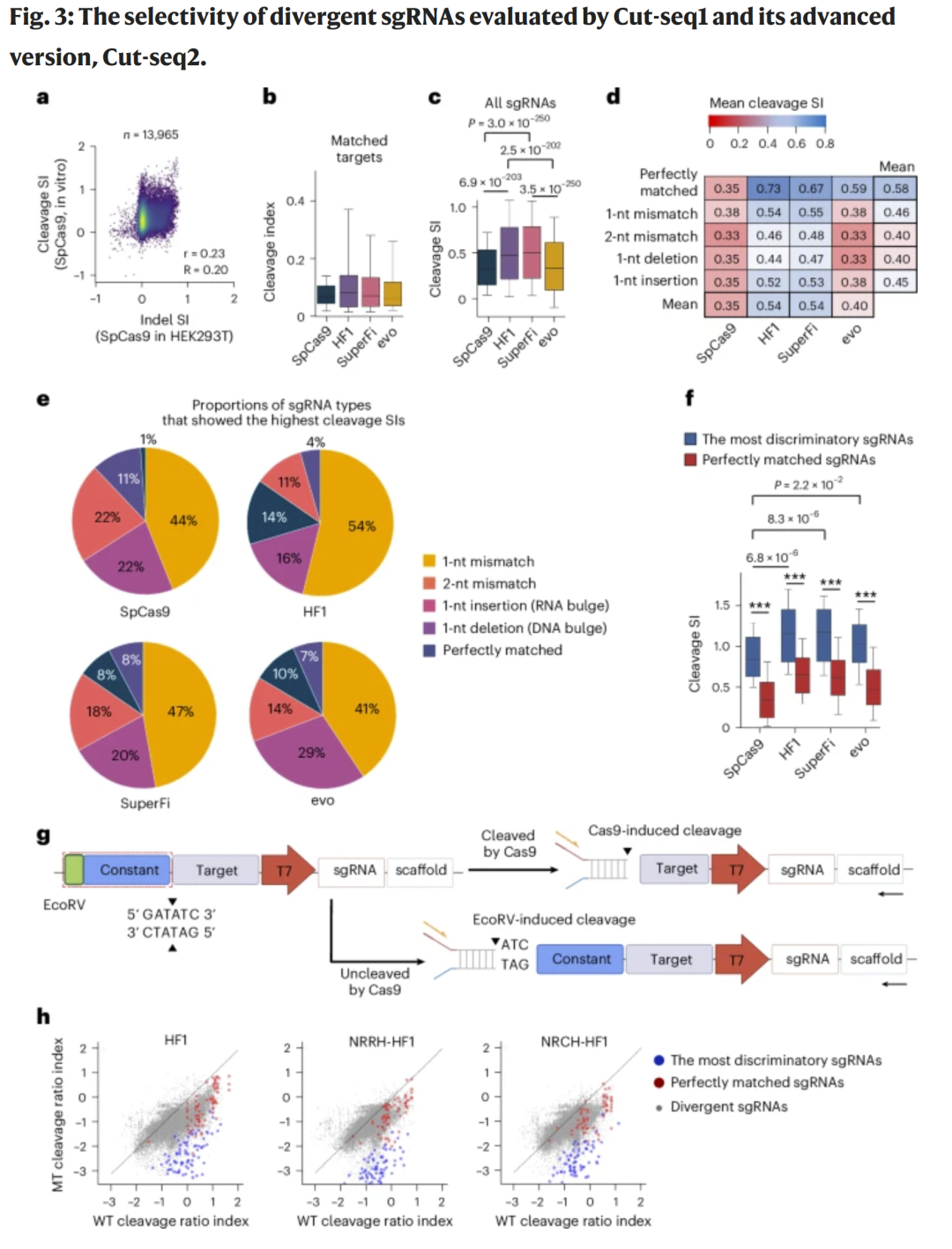
图3 差异 sgRNA 选择性及 Cut-seq2 方法
DeepCut 深度学习模型的建立与性能
利用 Cut-seq2 生成的 18 万余组 sgRNA–靶序列数据,研究人员开发了基于 Transformer 与卷积网络架构的 DeepCut-HF1、DeepCut-NRRH-HF1 与 DeepCut-NRCH-HF1 模型,用以预测切割比值指数。模型采用 sgRNA–靶序列配对编码及突变信息输入,显著优于传统机器学习方法,相关性高达 r = 0.90。

图4 DeepCut模型结构与性能评估
DeepCut 优化的 sgRNA 在稀有变体富集中的应用
研究人员利用 DeepCut 选取针对 2,612 个癌症突变的优化 sgRNA,构建了 optimized_HF1_2k 与 optimized_NRRH_2k 文库。通过多轮 Cas9 切割与 PCR 扩增(CLOVE-seq),研究人员在 7.8k 稀有变体文库中实现了高达 900 倍的平均等位基因频率提升,且超过 80% 的突变可被检测。相比之下,使用完全匹配 sgRNA 的富集效率明显较低。

图5 稀有变体多重富集效果
选择性机制与检测灵敏度分析
研究人员进一步分析了突变位置对选择性的影响,发现当突变位于 PAM 或 PAM 邻近的原间隔区时,富集效果最佳。此外,测序深度提高至 10,000× 后对检测灵敏度提升有限,说明该方法在标准深度下已具高稳定性。

图6 优化 sgRNA 介导的选择性切割与稀有变体检测策略示意
|| 讨论
研究人员通过 Cut-seq1 与 Cut-seq2 实现了迄今最大规模的体外 CRISPR 切割效率评估,并揭示体外活性与细胞内 indel 频率的差异来源于细胞环境复杂性。基于此构建的 DeepCut 模型不仅能准确预测体外切割效率,还能为 sgRNA 优化提供可解释性特征。结合 CLOVE-seq,研究人员实现了首个大规模稀有变体的高选择性、多重富集体系。该研究奠定了深度学习驱动的体外 CRISPR 优化与分子检测新范式,为未来临床基因变体检测、癌症早筛及个体化诊断提供了可扩展的技术基础。
参考资料
Yeo, J.H., Lee, S., Kim, S. et al. High-throughput evaluation of in vitro CRISPR activities enables optimized large-scale multiplex enrichment of rare variants. Nat. Biomed. Eng (2025).
https://doi.org/10.1038/s41551-025-01535-0
文章改编转载自微信公众号:DrugAI
原文链接:https://mp.weixin.qq.com/s/IuASIABlpjzMDiELsl0ejA | 






















