本帖最后由 哈奇一 于 2025-10-21 01:25 编辑
基于结构的分子生成(Structure-Based Molecular Generation, SBMG)代表了人工智能驱动药物设计的重要进展,然而该领域的发展受到蛋白–配体复合物结构数据稀缺的限制。浙江大学药学院侯廷军教授与谢昌谕教授团队、上海交通大学溥渊未来技术学院郑双佳助理教授团队以及徐州医科大学郭栋教授团队联合提出了一种新的分子生成方法EcloudGen。该方法通过引入电子云作为物理上有意义的潜在变量,连接仅含配体的数据与蛋白–配体复合物数据,从而使靶标感知的生成模型能够探索更广阔的化学空间。基于量子化学计算的启发,研究人员提出了ECloudGen模型,通过融合潜变量扩散模型、Llama架构与对比学习任务,将化学空间组织为结构化且可解释的潜在表征。基准实验显示,EcloudGen生成的分子相较于现有方法结合力更强、理化性质更优,且能覆盖更广泛的化学空间。电子云潜变量的引入不仅提升了生成性能,还赋予模型层面的可解释性,实验结果进一步验证了该方法的实用性。

药物研发是人类抗击疾病的关键途径之一。分子生成被视为人工智能驱动药物设计的“皇冠明珠”,目标是设计新颖且高效的候选药物。然而,早期分子图生成方法往往不具备靶点特异性,难以满足真实药物设计的需求。近年来,研究重点逐渐转向基于结构的分子生成(SBMG),即模型需根据蛋白结构特征生成具有特定结合能力的分子。
这一转变的本质是从建模 p(G) 过渡到建模 p(G|p),即以蛋白结构为条件预测分子。得益于扩散模型和大语言模型的崛起,研究人员能够处理更复杂的条件生成任务,从而催生出一系列 SBMG 模型,如 DiffBP、ResGen、Pocket2Mol 等。然而,这些方法普遍受到一个长期存在但常被忽视的问题限制——结构数据稀缺。相比于仅基于分子数据的生成任务可使用约 10¹⁰ 级别分子数据集,SBMG 仅能利用约 10⁵ 个蛋白–配体复合结构,导致生成模型在狭窄的化学空间中学习,难以探索新的分子构型。
研究人员将这一现象称为 “稀疏化学空间生成悖论”,并将其视为制约结构感知生成模型性能提升的核心瓶颈。
方法
为突破数据限制,研究人员开发了 ECloudGen 框架。其核心思想是引入潜在变量 𝐶,将生成过程分解为 p(G|C,p) × p(C|p)。这使得模型可以从配体–蛋白复合数据中学习分子生成模式,同时利用仅含配体的大规模数据来拓展潜空间。
与传统隐变量不同,ECloudGen 选择物理上具有明确意义的“电子云”作为潜变量。电子云不仅代表分子的量子特征,也是决定原子间相互作用的根本因素。这一选择在物理合理性、可解释性与可控性之间实现了平衡。
模型架构
ECloudGen 由两个主要模块组成:
ECloud Latent Diffusion 模块:
学习电子云的条件分布 p(C|p),在给定蛋白口袋结构的情况下生成高保真电子云分布。该模块基于三维潜变量扩散模型,能够在压缩潜空间内生成复杂的三维电子密度。
ECloud Decipher 模块:
学习 p(G|C,p),将生成的电子云与蛋白口袋信息共同解码为三维分子结构。此模块采用 Llama 架构中的 RoFormer 变体,并结合 3D-Unet 实现分子结构重建。
此外,研究人员设计了 CEMP(对比电子云预训练) 策略,通过对比学习使化学空间形成结构有序的潜在分布,从而提升模型的泛化与优化性能。
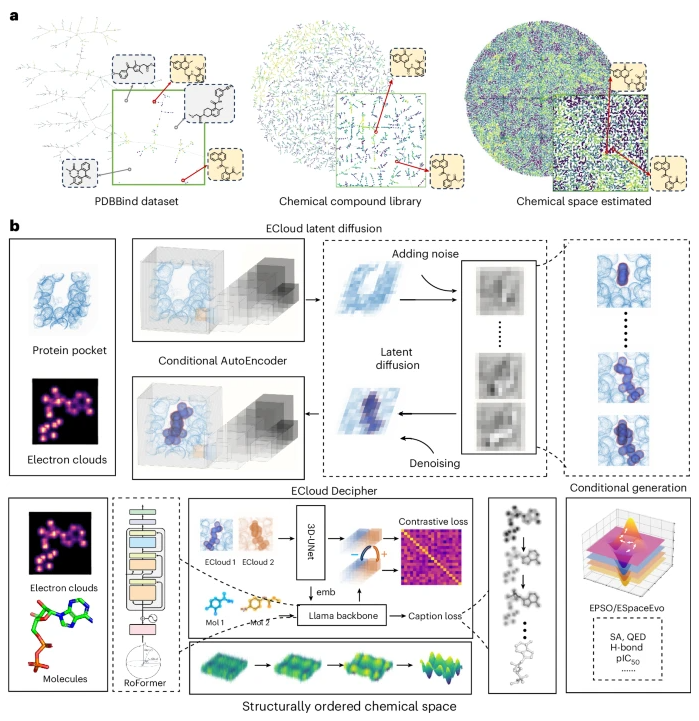
图1|稀疏化学空间悖论与 ECloudGen
结果
结合力与药物相似性
ECloudGen 与十种主流基线模型(Pkt2Mol、ResGen、GraphBP、FragGen、DiffBP、TargetDiff、Lingo3DMol 等)进行了系统比较。结果表明,ECloudGen 在 结合效率(LBE) 指标上表现最佳,说明其能生成结合更紧密的分子;在药物相似性(QED)与合成可及性(SA)方面同样表现优异。
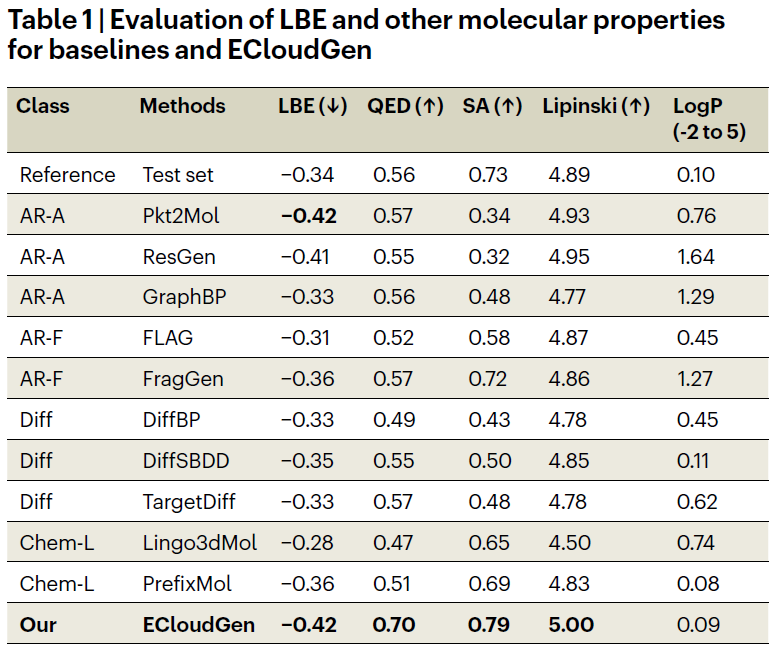
相比之下,扩散类模型虽然能生成高亲和性分子,但药物样性质偏弱;自回归模型在部分性质上改进明显,但受限于片段覆盖度。ECloudGen 则在所有指标间实现平衡。
化学空间覆盖与多样性
研究人员采用两种拓扑学度量方法——最大排斥圆(Circles) 与 瓶颈距离(Bottleneck),更精确地评估化学空间覆盖度。
ECloudGen 的 Circles 值达到 85,Bottleneck 值为 1,200,显著优于其他模型,表明其生成分子覆盖更广、分布更均衡。这归功于其通过电子云潜变量融合无结构配体数据,极大拓宽了化学空间。
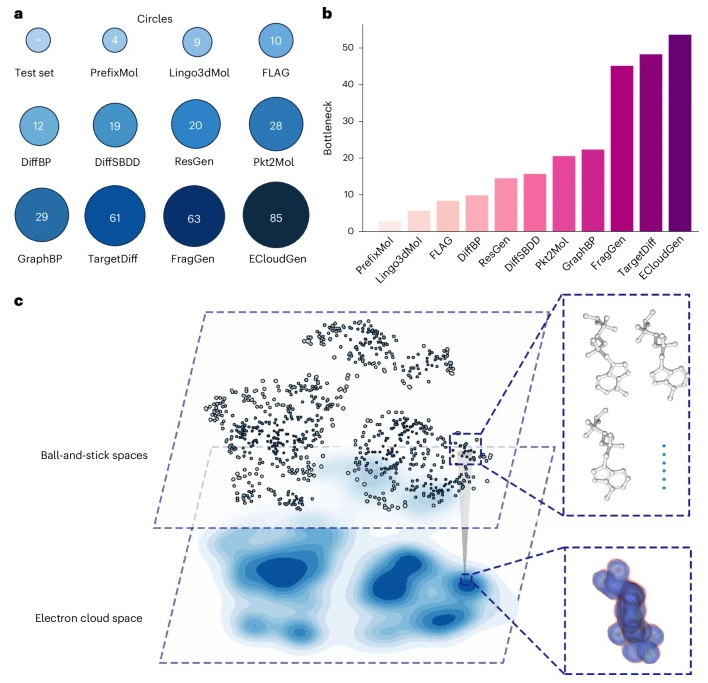
图2|化学空间度量与潜空间映射
分子优化性能
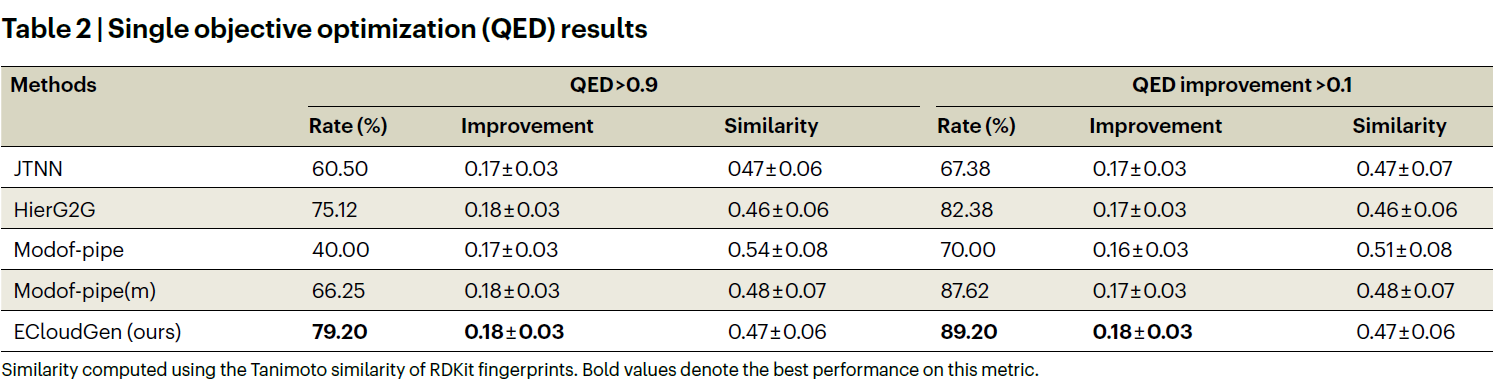
ECloudGen 进一步集成了两种分子优化算法:
ESpaceEvo:基于元动力学的连续优化;
EPSO:基于粒子群优化(PSO)的模型无关优化器。
以 QED 为目标进行优化时,ECloudGen 的成功率为 79.2%(QED>0.9),比次优模型高出约 5%,并能在无需额外训练代理模型的情况下实现高效优化。这一特性为分子设计提供了可插拔、可扩展的优化框架。
案例一:V2R 内源配体重设计
以血管加压素受体 V2R 为目标,研究人员通过电子云潜变量生成多个候选分布,并以 E-Confidence 分数 评估潜在活性。
选择高置信度电子云后,利用 ECloud Decipher 将其映射为具体分子结构。合成并测试的两种候选分子均表现出微摩尔级活性,验证了 ECloudGen 在受体结合位点重设计中的实际效用。
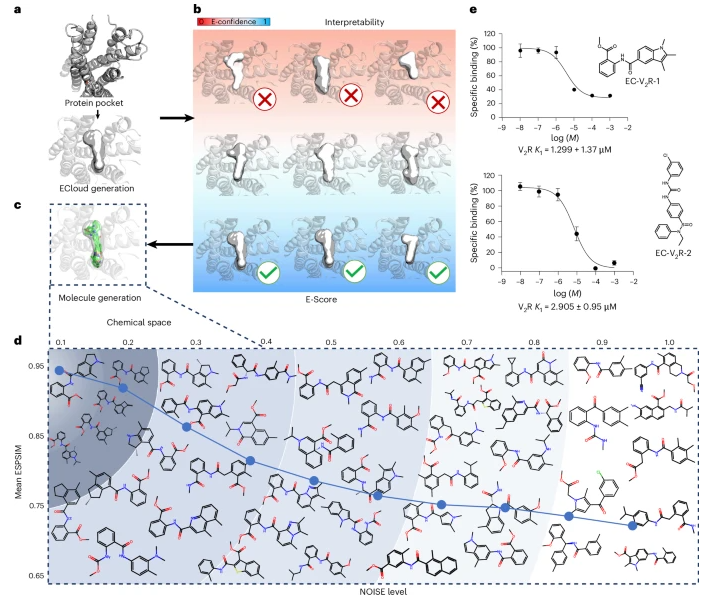
图3|V2R 受体配体重设计流程
案例二:BRD4 多目标优化
针对肿瘤相关靶点 BRD4,研究人员以内源配体乙酰化赖氨酸为起点,进行多目标分子优化。优化目标包括 QED、SA、分子量、Lipinski 法则及氢键受体数。经过 50 轮优化后,大多数分子满足五项约束。分子对接结果显示优化分子保留关键氢键(Asn140 与 Tyr97),两种被选中合成的分子在生物测定中表现出 25 μM 和 0.5 μM 的抑制活性,展示了 ECloudGen 在多目标药物优化中的可行性。
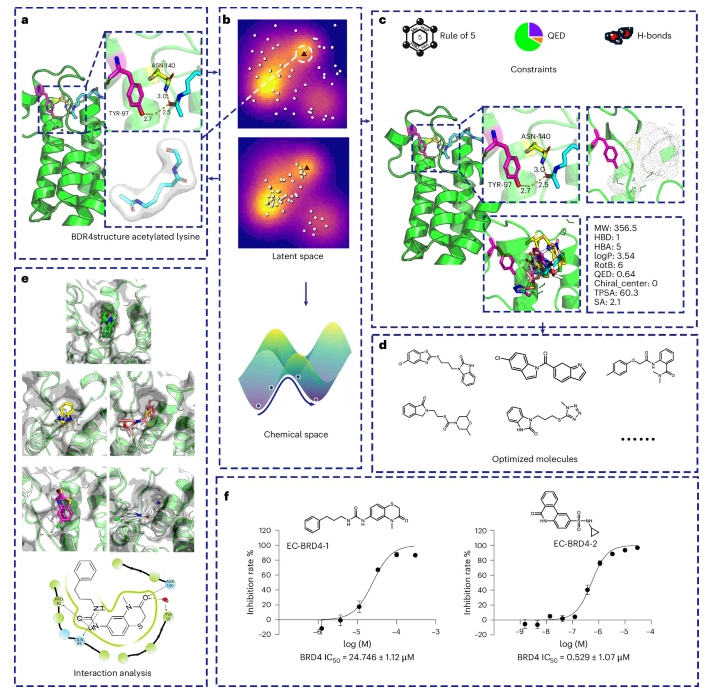
图4|BRD4 多目标优化流程
讨论
ECloudGen 通过引入电子云潜变量,从根本上缓解了 SBMG 中的稀疏化学空间问题。其将蛋白–配体对与仅含配体数据桥接,结合物理意义与生成建模优势,实现了化学空间的结构化组织与模型级可解释性。
该方法的主要瓶颈在于电子云生成阶段,其精度受限于可用蛋白–配体数据的质量与数量。未来可通过更强的三维生成模型或基于表面生成的策略优化这一环节。ECloudGen 的设计理念还可扩展至蛋白设计、材料生成等其他分子生成任务,为物理可解释的生成式人工智能提供新方向。
参考资料
Zhang, O., Jin, J., Wu, Z. et al. ECloudGen: leveraging electron clouds as a latent variable to scale up structure-based molecular design. Nat Comput Sci (2025).
https://doi.org/10.1038/s43588-025-00886-7
整理 | 王建民
文章改编转载自微信公众号:DrugAI
原文链接:https://mp.weixin.qq.com/s/al36jgAVr_8NpxjVDvjI7Q?scene=1 | 








