本帖最后由 薛定谔了么 于 2025-12-24 23:30 编辑
2025年11月25日,复旦大学韩涟漪、夏晶晶团队在《Communications Biology》期刊上发表研究论文,题为“MoleculeFormer is a GCN-transformer architecture for molecular property prediction”。该研究提出了一种基于GCN-Transformer架构的多尺度特征融合模型MoleculeFormer。在覆盖28个数据集上的实验结果表明,该模型在多种药物发现任务中均表现出稳健性能,包括药物疗效/毒性预测、表型筛选以及ADME评估。

MoleculeFormer代码仓库:https://github.com/qinmingyuan-hub/molecular_graph
背景
近年来,基于人工智能的方法在小分子性质预测的准确性与计算效率方面取得显著进展。这类方法依托机器学习与深度学习技术,能够更深入地刻画分子结构与理化性质及生物活性之间的内在关联,从而实现对分子生物活性的有效预测。
在此过程中,小分子特征嵌入仍是至关重要的环节,高效、充分地提取与任务相关的分子特征被视为当前研究的核心方向之一。现有方法主要涵盖分子表示与原子表示两种范式。分子表示通常基于先验化学知识对整体分子特征进行编码,典型形式包括分子描述符和分子指纹;而原子表示则从原子层面出发,将SMILES表达式转换为图结构,其中原子被建模为节点、化学键被建模为边,并对原子及化学键的属性(如原子序数、价电子数以及单键或双键类型等)进行特征嵌入。通过聚合邻域节点信息并迭代更新节点表示,模型能够有效捕获分子的拓扑与结构特征。实践表明,将基于图的原子表示与基于描述符的分子表示进行融合,通常可以进一步提升模型的预测性能与泛化能力。
方法
本研究提出了一种基于多尺度特征的可解释分子预测模型MoleculeFormer。在图编码策略上,不同于传统仅采用单一分子图表示的方法,模型引入了特征维度为39的键图信息。具体而言,将每一条化学键视为一个节点,并根据化学结构关系连接相邻化学键。相较于主要关注原子属性的原子图表示,键图能够作为其重要补充,更为精细地刻画原子之间的成键关系,包括键类型、键长及键角等关键信息,从而增强对分子局部结构的表征能力。
在图特征聚合方面,传统图模型通常采用节点级聚合机制,通过节点与其邻域之间的信息传递,并最终借助最大池化或平均池化等操作获得图级表示。然而,此类方法往往伴随较为严重的信息损失,且池化过程本身在模型层面缺乏可解释性。受自然语言处理领域相关思想的启发,研究团队采用GCN模型替代位置编码,并引入Transformer编码器,将分子图整体视作一个句子进行特征建模。通过分析图表示节点与各节点之间的相关性,实现了对整个图特征的聚合与聚类。

图1 MoleculeFormer整体架构
在表示与聚合阶段,模型进一步引入三维特征进行编码,并融合等变图神经网络(EGNN),以保持对旋转和平移变换的等变性。在满足图等变约束的同时,该设计构建了一个多维度、具有良好可解释性的建模框架。借助注意力机制,并结合原子图与键图的分子特征提取过程,模型能够在微观层面实现分子结构注意力的可视化表达。此外,模型还引入包含先验化学知识的分子指纹编码,以提升预测精度并加快模型收敛速度(图1)。整体而言,该多维分子性质建模框架能够充分挖掘分子结构信息,从而保障其在实际应用任务中的可靠性与预测准确性。
结果
分子指纹组合的选择
实验结果(图2)表明,不同单一分子指纹在不同预测任务中的性能差异显著。在分类任务中,ECFP指纹和RDKit指纹表现最为突出。在回归任务中,MACCS keys指纹表现最佳。值得注意的是,EState指纹在单一分子指纹设置下整体表现相对较弱,在分类和回归任务中分别排名第四和最后,表现均劣于其他指纹;然而,在组合使用时却展现出优异性能,这反映了指纹特征互补性的重要作用。此外,RDKit指纹在回归任务中表现出一定的干扰效应,对模型性能产生负面影响。
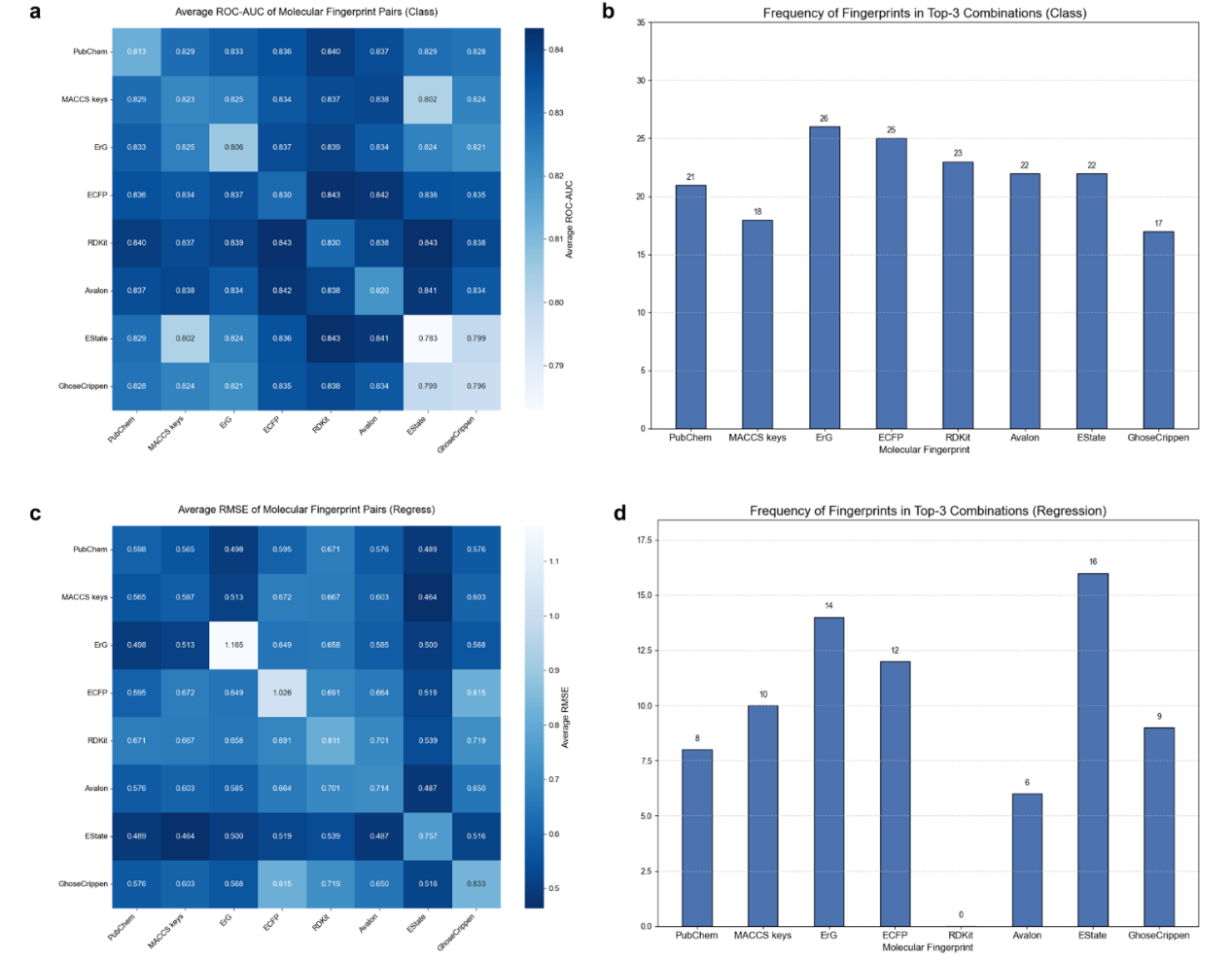
图2 分子指纹选择实验结果
在双指纹组合实验中,不同任务对应的最优组合亦存在明显差异。在分类任务中,ECFP指纹与RDKit指纹的组合取得了最优性能,表明二者在分类任务所需的特征表达方面具有较强的互补性,其信息融合有助于模型更准确地区分分子类别。性能相当的组合还包括EState指纹与RDKit指纹。在回归任务中,MACCS keys与EState指纹的组合表现最为优异。
进一步地,在跨不同数据集评估平均性能最优的三指纹组合时,在分类任务中,各模型整体表现较为均衡,最优组合为ErG、ECFP和RDKit指纹。在回归任务中,EState指纹表现最为突出,而RDKit指纹表现较差,未进入排名。因此,在回归模型的最终设置中,作者选择了EState指纹、ErG指纹和ECFP指纹作为分子指纹输入。
性能比较
本研究采用Wu等人提出的公共基准数据集,对MoleculeFormer在10个广泛使用的药物性质预测基准数据集上的性能进行了系统评估。如表1所示,MoleculeFormer在13项任务中取得了最佳的平均性能,充分验证了其有效性。
表1 在10个常用公共数据集上的预测性能结果

在具体数据集上,MoleculeFormer在BACE和BBBP数据集的二分类任务中表现出色。在ClinTox数据集上的平均PR-AUC为0.98±0.01。相比之下,在SIDER、HIV、Tox21以及MUV数据集上,PR-AUC值相对较低。这表明这些数据集上的整体分类难度较高,可能存在样本类别不平衡以及特征复杂多样等问题。尽管如此,与现有对比模型相比,MoleculeFormer仍然展现出更为显著的性能优势。
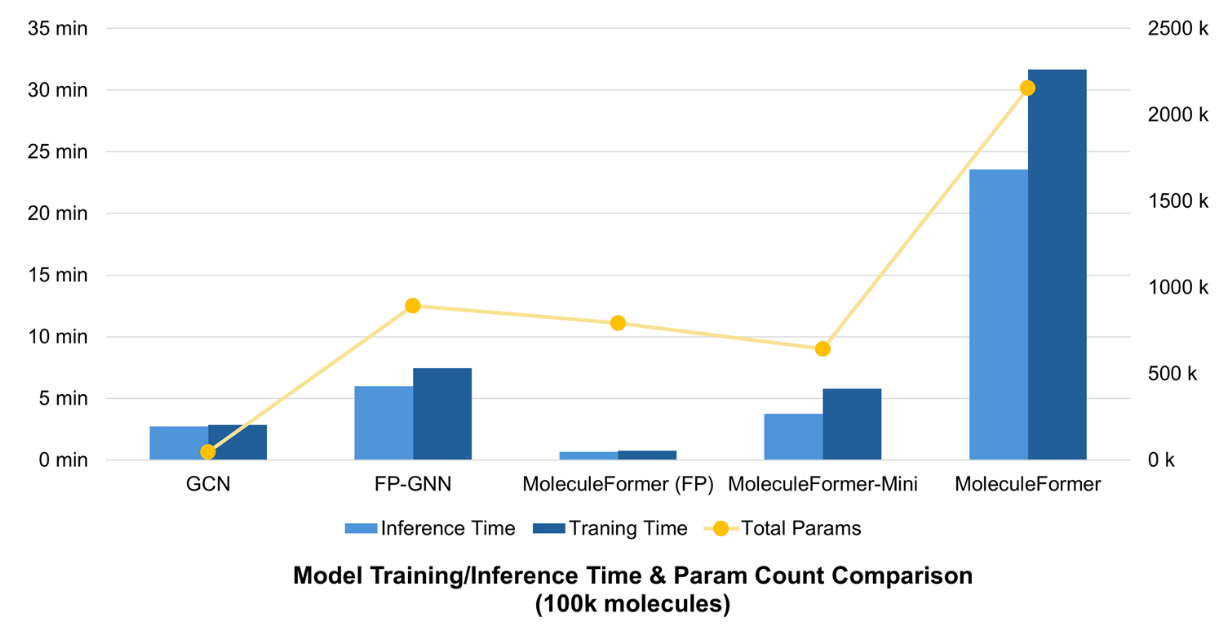
图3 训练/推理时间与参数规模对比
图3展示了在输入10万个包含三维结构信息的小分子后,不同模型在训练与推理阶段所需的时间以及对应的参数规模。MoleculeFormer(FP)直接利用分子指纹进行全连接编码,因此其训练和推理速度相对较快。MoleculeFormer-Mini版本进一步移除了EGNN模块,在显著降低训练时间的同时,保留了GCN与Transformer的核心结构,从而在效率与性能之间取得了较好的平衡。
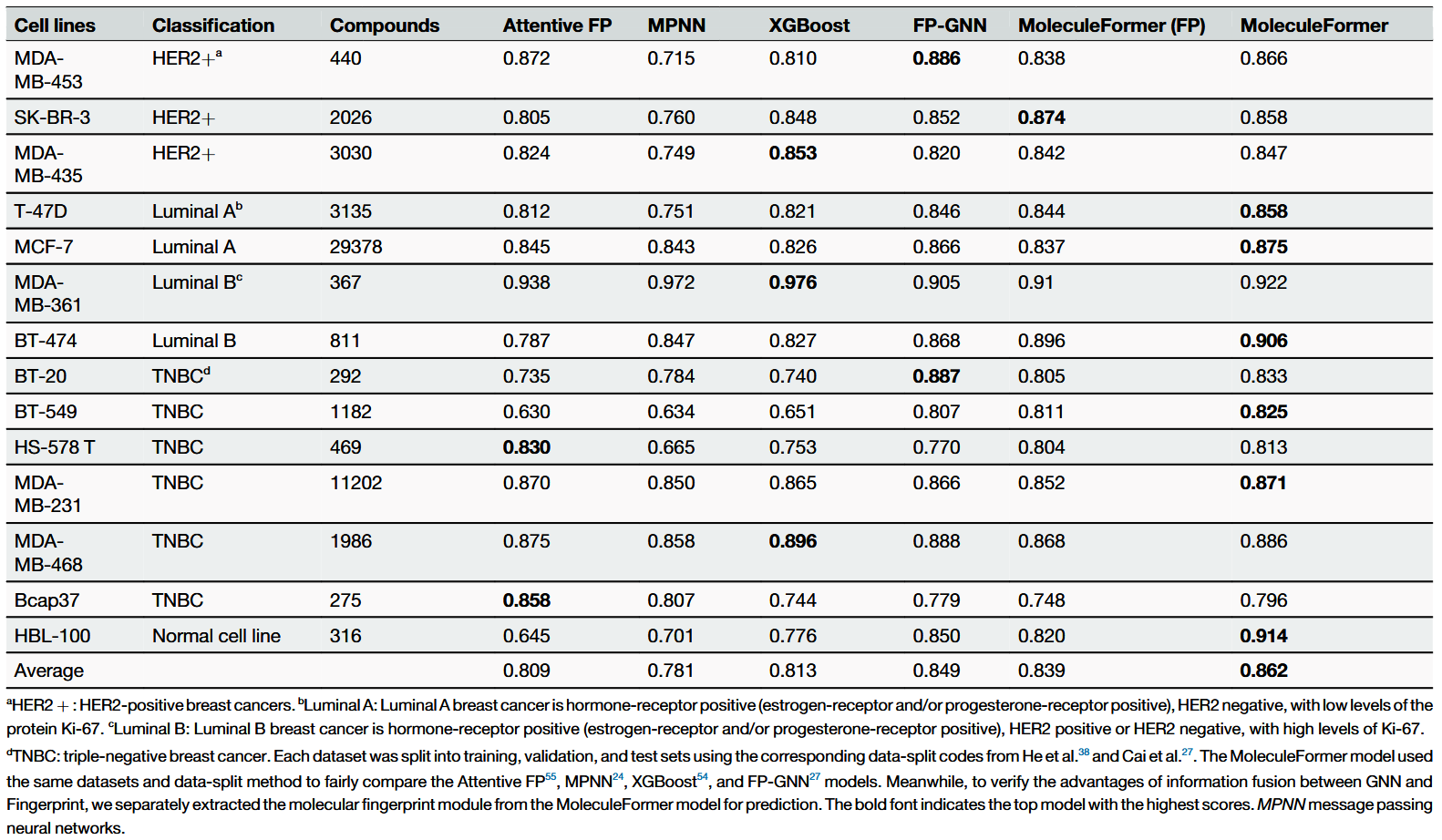
为评估MoleculeFormer是否能够捕获药物的整体效应,并有效反映其对真实细胞体系的综合影响,研究进一步在一个化合物–乳腺癌细胞表型筛选数据集上开展了实验。如表2所示,各细胞系中性能最优的模型结果以粗体标示。MoleculeFormer在所有细胞系上的平均ROC-AUC得分均为最高,表明其在细胞表型预测任务中具备极具竞争力的预测精度。
表2 在14种乳腺细胞系数据集上的ROC-AUC预测性能结果
消融实验
本研究选用的Biogen数据集主要聚焦于已上市化合物的ADME特性,涵盖结构高度多样化的分子,能够较为真实地反映候选药物在实际研发场景中的复杂性。在消融实验中,系统比较了六种模型架构:基线图卷积神经网络(GCN)、基于原子图的Transformer模型(Atom)、原子图与键图联合的Transformer模型(Atom+Bond)、分子指纹编码模型(FP)、融合分子指纹的原子图与键图联合Transformer模型(Atom+Bond+FP),以及同时集成原子图、键图、分子指纹和EGNN模块的复合模型,即MoleculeFormer。
表3 在4个不同数据集上的消融实验结果
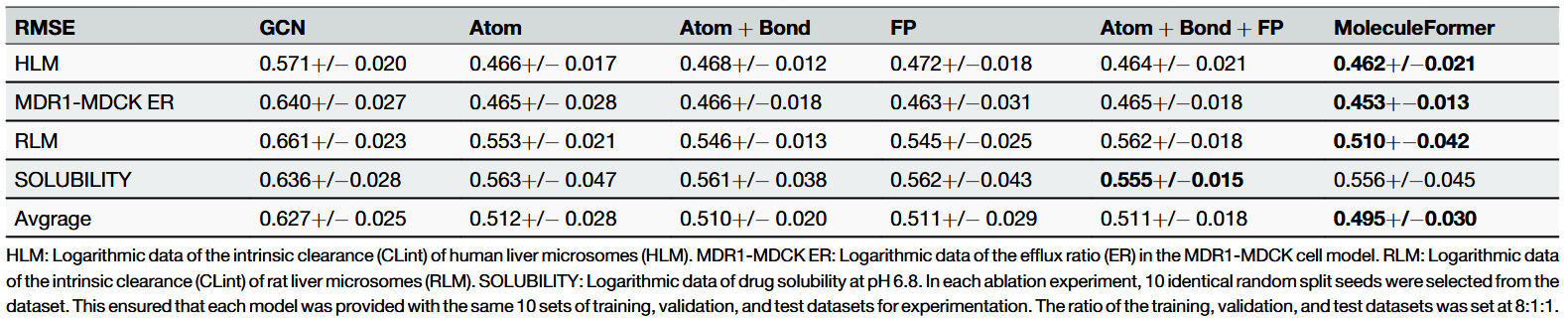
如表3所示,消融实验结果可归纳为以下三点主要结论:(1)原子图与键图的协同作用:将原子图与键图进行融合,能够有效增强对隐式分子结构特征的建模能力,并显著提升预测结果的稳定性。(2)多模态特征的互补性:引入分子指纹后,模型的整体表征能力得到明显提升,其与基于图的结构化特征形成良好互补,从而构建更加全面、信息丰富的分子表示。(3)三维特征敏感性:在涉及分子构象或空间依赖关系的数据集中,引入EGNN模块能够显著提升模型性能,充分体现了其在捕获三维几何结构特征方面的优势。
抗噪声能力
在HIV数据集上,研究对MoleculeFormer的抗噪声能力进行了系统评估。具体而言,在训练集和验证集中人为反转一定比例的标签,并在10个不同的随机种子下重复实验。图4的结果表明,随着噪声比例的增加,MoleculeFormer依然能够保持相对稳定的性能,展现出卓越的抗噪声鲁棒性,说明其在低质量数据集条件下仍能保持良好的性能。
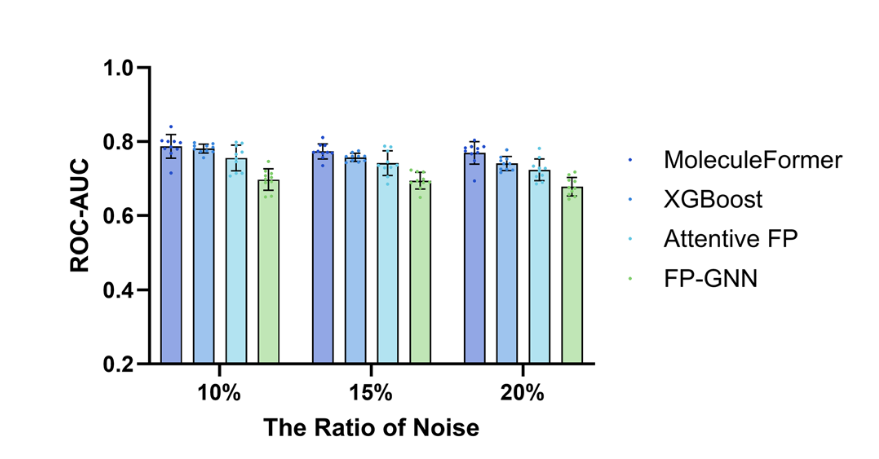
图4 不同噪声比例下的抗噪声性能对比
脂溶性与氢键供体能力分析
为进一步探究模型的可解释性,每个分子被划分为两个结构片段,并分别计算其cLogP值。同时,对Transformer层中的注意力权重进行可视化分析,其中颜色深浅表示各原子所分配的注意力权重,颜色越深代表权重越高。结果显示,模型在cLogP值较高的区域分配了更多注意力(图5a)。此外,键图表示更倾向于关注分子中的极性基团,从而对氢键供体(HBD)特性产生显著影响;相较之下,碳链结构通常具有较低的三维刚性,更有利于分子通过血脑屏障(BBB)的孔径限制(图5b)。
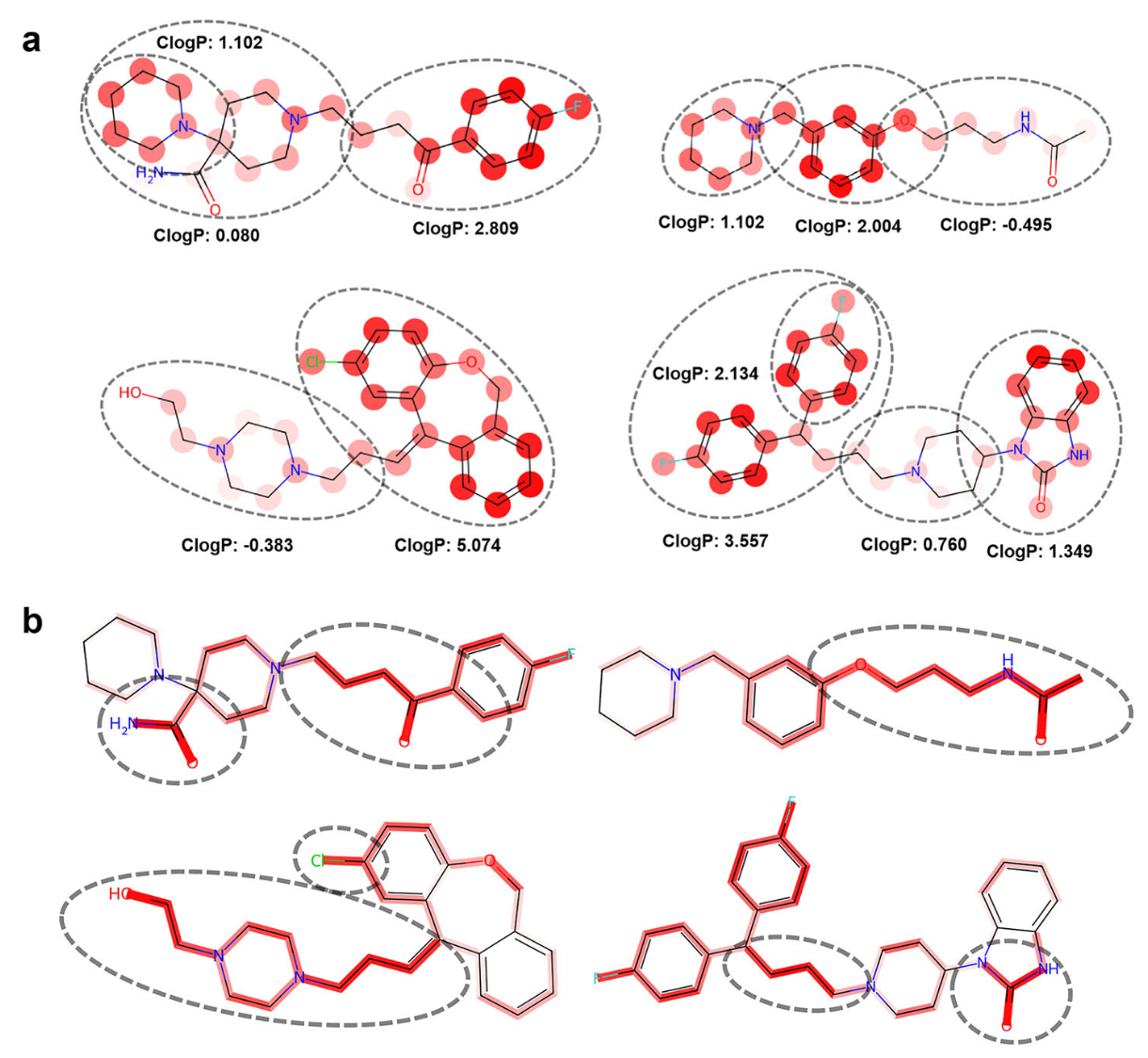
图5 Transformer层中注意力权重的可视化结果
在具体案例分析中,Brand等人对BBB渗透性较差的化合物1进行了两轮优化,较高的Kp和cLogP值表示更强的BBB渗透能力(图6a)。Fushimi等人对BBB渗透性较差的化合物4同样进行了两轮优化。MDR1(P-糖蛋白)是BBB中的一种转运蛋白,能够主动将药物外排,从而降低其进入大脑的能力,MDR1水平越低,BBB渗透性越强(图6b)。Gunaga等人通过结构修饰降低了原本具有较高BBB穿透风险的化合物7的风险,较低的C_brain/C_plasma比值表示更弱的BBB渗透能力(图6c)。在上述案例中,MoleculeFormer的预测结果均与实际实验结果保持一致。
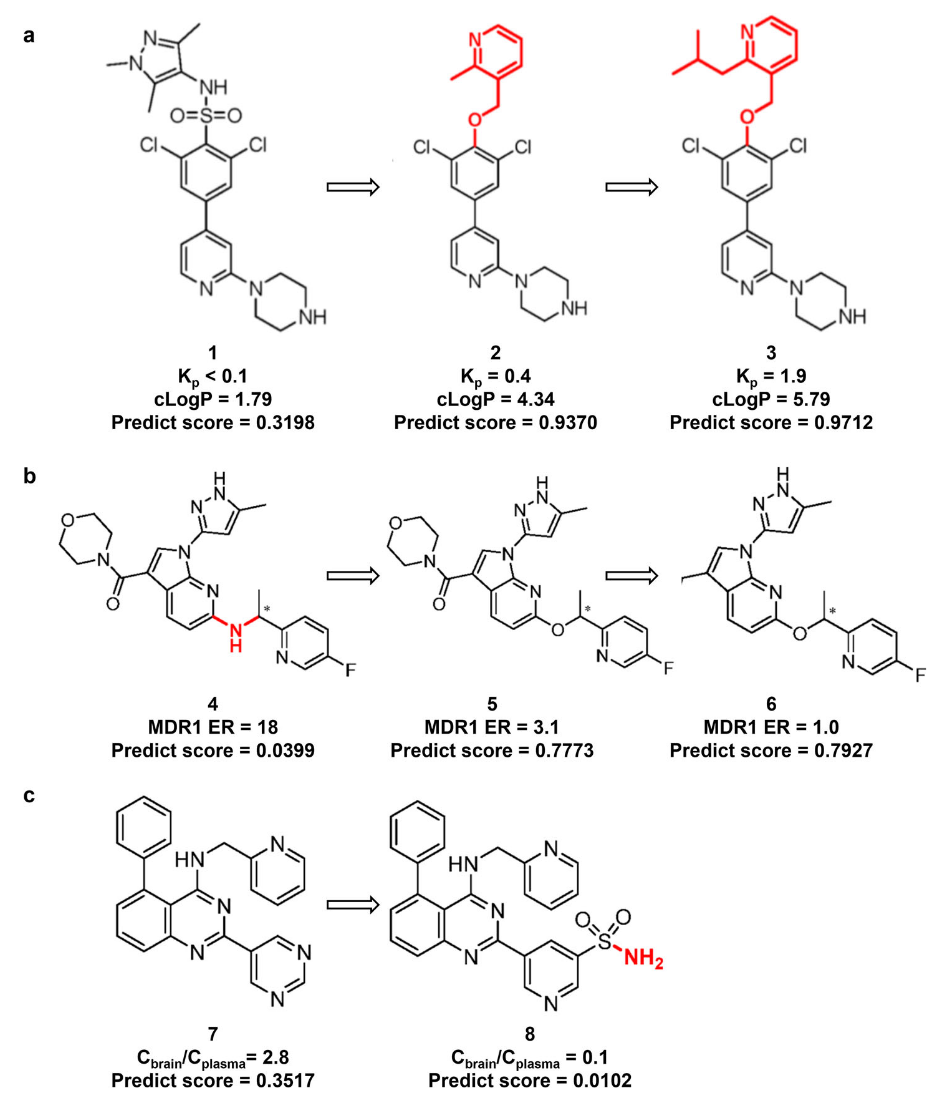
图6 化合物BBB渗透性优化过程及模型预测一致性的验证
综合上述结果可以看出,MoleculeFormer具备较强的可解释性,能够有效识别并量化决定BBB通透性的关键分子特征,如脂溶性和氢键供体能力,且这些特征与经典的药物化学理论高度一致。这种可解释性使该模型在实际应用中具有重要价值。不仅能够预测BBB渗透能力,还可提供符合化学直觉的结构层面解释,从而为中枢神经系统药物的理性设计提供直接指导。
总结
本研究提出了一种基于多尺度特征的可解释分子性质预测模型。该模型以GCN–Transformer为核心架构,分别从原子图和键级图两个层面提取分子特征:原子图通道通过全局注意力机制动态建模分子内部原子之间的长程相互作用,而键图通道则重点刻画局部化学键的拓扑结构与电子效应。二者的协同建模不仅显著增强了模型对分子多层次特征的解析能力,还能够借助注意力权重的可视化揭示潜在的作用机制,例如分子预测任务中的关键官能团及隐含结构特征。此外,模型在设计中同时引入了分子图的旋转等变性约束以及分子指纹所蕴含的先验化学知识,使其在局部与全局分子特征的联合建模方面表现尤为出色。
为全面评估模型的通用性与可靠性,研究系统性地选取了覆盖药物研发关键阶段的多维数据集,包括10个MoleculeNet核心子集、14个表型筛选数据集以及ADME药代动力学数据集。对比实验与消融实验结果表明,该模型能够以互补方式有效整合多种编码范式,在跨领域、多任务的分子预测场景中均展现出稳定且优异的性能,充分验证了其在复杂分子表征与多目标预测任务中的泛化能力。
参考链接:
https://doi.org/10.1038/s42003-025-09064-x
文章改编转载自微信公众号:智药邦
原文链接:https://mp.weixin.qq.com/s/Pf5mVRV0c07j3RGxwhLc5A | 


























