本帖最后由 离子 于 2025-6-5 18:32 编辑
《Deep-learning-based single-domain and multidomain protein structure prediction with D-I-TASSER》一文提出了融合深度学习与物理势的片段组装结构预测方法 D-I-TASSER,可实现原子级精度蛋白质结构建模,还能自动预测大型多结构域蛋白质。基准测试和 CASP15 盲测显示,其精度优于 AlphaFold2 和 AlphaFold3,且在 CASP15 两项任务中均获冠军。该研究为蛋白质结构预测及动力学研究提供新方向。从技术交叉看,其算法思路或为量子计算机模拟复杂物理系统提供建模参考,与相干光量子计算机结合或助药物研发,长远看其深度学习模块或可迁移至量子计算框架。
2025年5月23日,新加坡国立大学张阳教授团队在《Nature Biotechnology》上发表了题为《Deep-learning-based single-domain and multidomain protein structure prediction with D-I-TASSER》(基于 D-I-TASSER 的深度学习单结构域和多结构域蛋白质结构预测)的论文。

这项研究工作提出了一种融合深度学习与物理势的片段组装结构预测方法D-I-TASSER,用于原子级精度的蛋白质结构建模。D-I-TASSER还引入了结构域划分与组装模块,实现了大型多结构域蛋白质的自动预测。大规模基准测试和CASP盲测结果显示,D-I-TASSER在单结构域与多结构域蛋白质结构预测问题中,精度均优于DeepMind公司的诺奖算法AlphaFold2和AlphaFold3。此外,在最近举办第15届世界蛋白质结构预测大赛(CASP15)中,D-I-TASSER算法在单结构域和多结构域蛋白质结构预测任务中均获得冠军。
本研究的第一作者为密歇根大学郑伟博士(现任南开大学统计与数据科学学院教授),共同第一作者包括密歇根州立大学乌云其其格博士与新加坡国立大学李阳博士(现为特别研究员)。通讯作者为密歇根大学Lydia Freddolino副教授和新加坡国立大学张阳教授。
一、研究思路
蛋白质三维结构预测(也称“蛋白质三级结构预测”)是指基于氨基酸序列推测其三维空间结构的过程。由于蛋白质只有在折叠成特定的空间构象后才能具备相应的活性和生物学功能,因此结构预测对于理解蛋白质功能、揭示疾病机制以及进行药物设计具有重要意义。传统的结构解析依赖于X射线晶体学、核磁共振等实验技术,不仅耗时长、成本高,还对实验条件要求苛刻。
近年来,该领域的一个重要里程碑是使用深度学习预测空间几何约束结构特征,例如接触图(contact map)、距离图(distance map)、氢键网络(hydrogen bond network)和扭转/二面角(torsion/dihedral angle),然后通过构建几何约束的能量函数,并采用能量最小化策略,进而建立蛋白质全长结构的预测模型。随后,AlphaFold2引入了端到端学习框架,直接根据序列和共进化信息预测蛋白质的结构,极大地提高了预测精度和效率。最新的AlphaFold3结合扩散模型,实现了另一种端到端结构预测方法。这一系列深度学习方法的出现,使得蛋白质结构预测准确率远远超过传统的基于物理势的结构预测方法(例如I-TASSER、Rosetta和QUARK)。然而,传统的基于物理势的结构预测方法在揭示蛋白质折叠原理与动力学机制方面具有不可替代的重要价值。在实际结构预测任务中,深度学习方法能否完全取代传统物理建模手段,仍有待进一步探讨。
目前蛋白质结构预测问题仍面临另一个重要挑战,即大多数先进方法主要聚焦于单个结构域(single domain)的预测。结构域(domain)是复杂蛋白质三级结构中的基本折叠和功能单元,大约60%的原核生物蛋白质和80%的真核生物蛋白质都由多个结构域(multi-domain)组成,并且依赖结构域间的相互作用执行更复杂的生物学功能,因此在结构上更为复杂(图1)。目前,无论是基于物理的结构预测方法,还是基于深度学习的结构预测方法,在处理多结构域蛋白质结构预测问题时都缺乏有针对性的多结构域处理模块。因此,如何准确高效准确地预测多结构域蛋白质结构,仍然是亟需解决的关键科学问题。
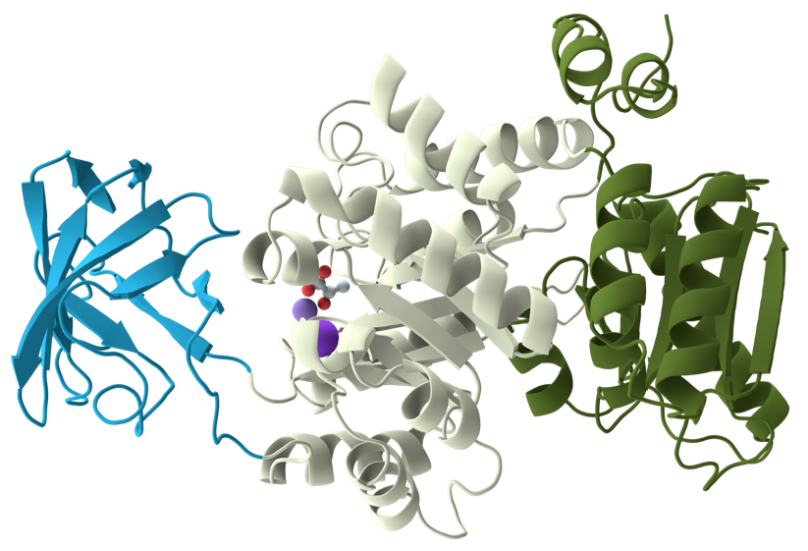 图1 多结构域蛋白质单体示意图 图1 多结构域蛋白质单体示意图
该蛋白质包含3个独立的结构域,不同结构域被标记为不同颜色。
在这项研究中,张阳教授团队在其以往开发的基于物理势的I-TASSER框架的基础上提出了一种整合深度学习的预测方法D-I-TASSER。D-I-TASSER结合了深度学习与物理势,采用副本交换蒙特卡洛模拟(REMC)进行蛋白质结构预测。同时,引入了一个全新的结构域划分与组装模块,用于自动预测大型多结构域蛋白质结构。基准测试和CASP15盲测结果均表明,D-I-TASSER在结构预测的准确性上明显优于传统的I-TASSER系列方法,以及Google公司DeepMind团队开发的AlphaFold2、AlphaFold3算法。
二、算法简介
D-I-TASSER算法能够有效处理单结构域蛋白质和多结构域蛋白质结构预测问题(图2)。对于单结构域蛋白质,D-I-TASSER首先使用DeepMSA2深度序列比对算法,迭代搜索基因组(UniProt、UniRef等)和宏基因组(Metaclust、IMG/M,BFD等)蛋白质序列数据库,构建深度多序列比对(MSA)。随后,利用LOMETS3模板检索算法,快速识别与目标蛋白结构相似的模板,并生成相应的模板比对。接着,使用DeepPotential、AttentionPotential、AlphaFold2等深度学习模块预测接触图(contact map)、距离图(distance map)、氢键网络(hydrogen bond network)等空间约束。最后,在深度学习空间约束与物理势共同构成的力场驱动下,采用REMC蒙特卡洛模拟算法进行蛋白质结构进行组装、聚类和优化。
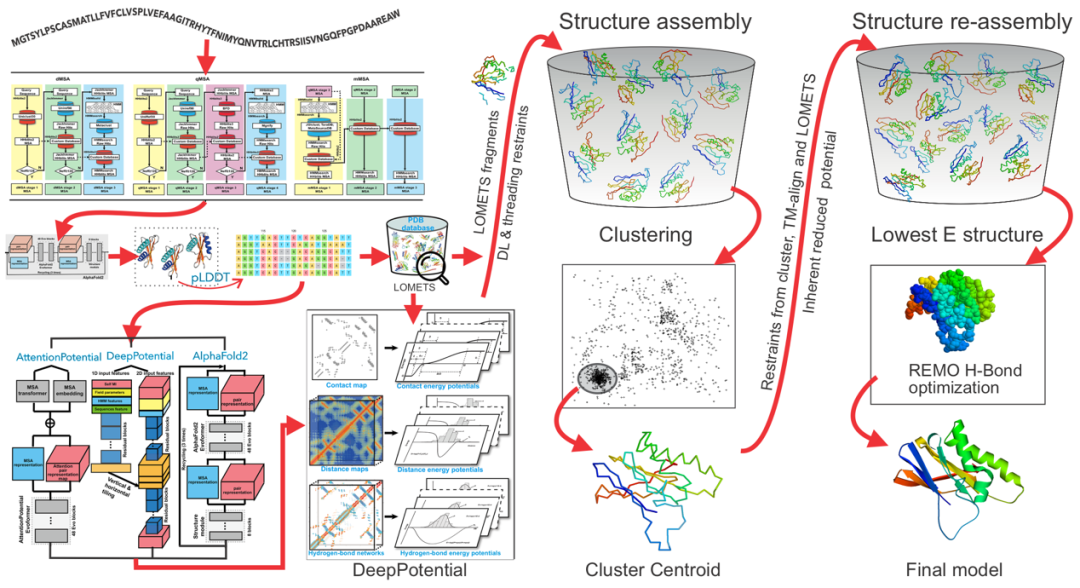 图2 D-I-TASSER单结构域蛋白质结构预测流程 图2 D-I-TASSER单结构域蛋白质结构预测流程
对于多结构域蛋白质,D-I-TASSER能够成功地预测其结构,得益于其引入了一个全新的结构域划分与组装模块(图3),该模块包含5个关键步骤:
预测结构域边界;
生成结构域级特征;
构建全链MSA与空间约束;
收集全链模板;
全链级结构预测。
具体地,首先使用ThreaDom和FUpred预测目标蛋白质的结构域边界,并将目标蛋白质划分为多个结构域。随后,为每个结构域生成结构域级的MSA、模板和空间约束。在此基础上,将原始的全链MSA与结构域级MSA结合,通过棋盘式对齐方式构建全链级MSA。将全链的MSA输入至深度学习模块,以获取全链级别的空间约束,并进一步与各结构域预测所得的空间约束信息融合,最终构建出完整的全链空间约束体系。全链级模板则包含DEMO2将各结构域模板拼接成的覆盖全链的模板,以及全链级LOMETS3模板。最后,通过全链的D-I-TASSER组装模拟,在混合的结构域级和跨域空间约束的引导下,构建多域结构模型。
D-I-TASSER与以往的多结构域结构预测算法I-TASSER-MTD的不同之处在于,之前的算法尝试将多结构域蛋白质分割后独立预测每个结构域的结构,然后再进行拼装。这样做的缺点是结构域内约束和结构域间约束可能会冲突,导致预测结果不稳定。D-I-TASSER采用的是结构域级的特征拼装,再将拼装好的完整的全链级的特征输入到深度学习模型、模板检索算法或者结构预测模块,进行整体预测,这样能够最大程度地减少结构域内约束和结构域间约束的冲突,提升预测精度。
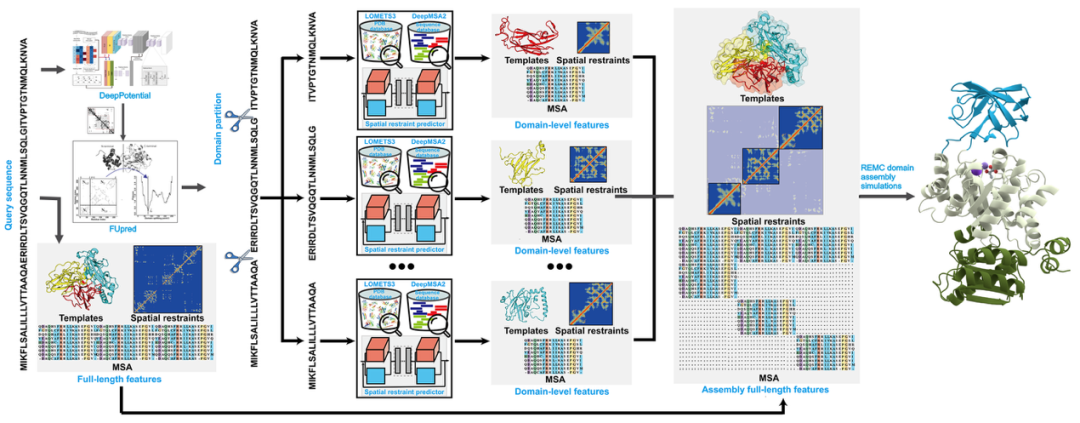 图3 D-I-TASSER多结构域蛋白质结构预测流程 图3 D-I-TASSER多结构域蛋白质结构预测流程
三、结果分析
D-I-TASSER在单结构域蛋白质结构预测任务中表现出显著领先的整体精度,全面优于其先代算法I-TASSER及AlphaFold各系列算法。在500个非冗余的“困难”结构域测试集上(困难蛋白质是指在PDB库中找不到有效同源结构模板的蛋白质),D-I-TASSER的平均TM-score达到0.870,相比最新版AlphaFold2的精度(TM-score=0.829)提升了5.0%,且在84%的目标上生成了质量更高的预测结果。对于特别困难的148个结构域(图4红色标注区域),D-I-TASSER表现大幅优于AlphaFold2(TM-score=0.707 vs 0.598)。其中,D-I-TASSER在63个蛋白质目标上的预测精度优于AlphaFold2,且差异超过0.1分;而AlphaFold2仅在8个目标上展现出相对优势。在更大规模的基准测试中(图5),D-I-TASSER的预测性能同样优于所有版本的AlphaFold,再次印证了其在单结构域蛋白质结构预测中的卓越性能。
注:TM-score是由张阳实验室首先提出、现已成为蛋白质结构相似性评估的主流指标。该分数范围在0到1之间,值越高表示预测结构与真实结构越接近。
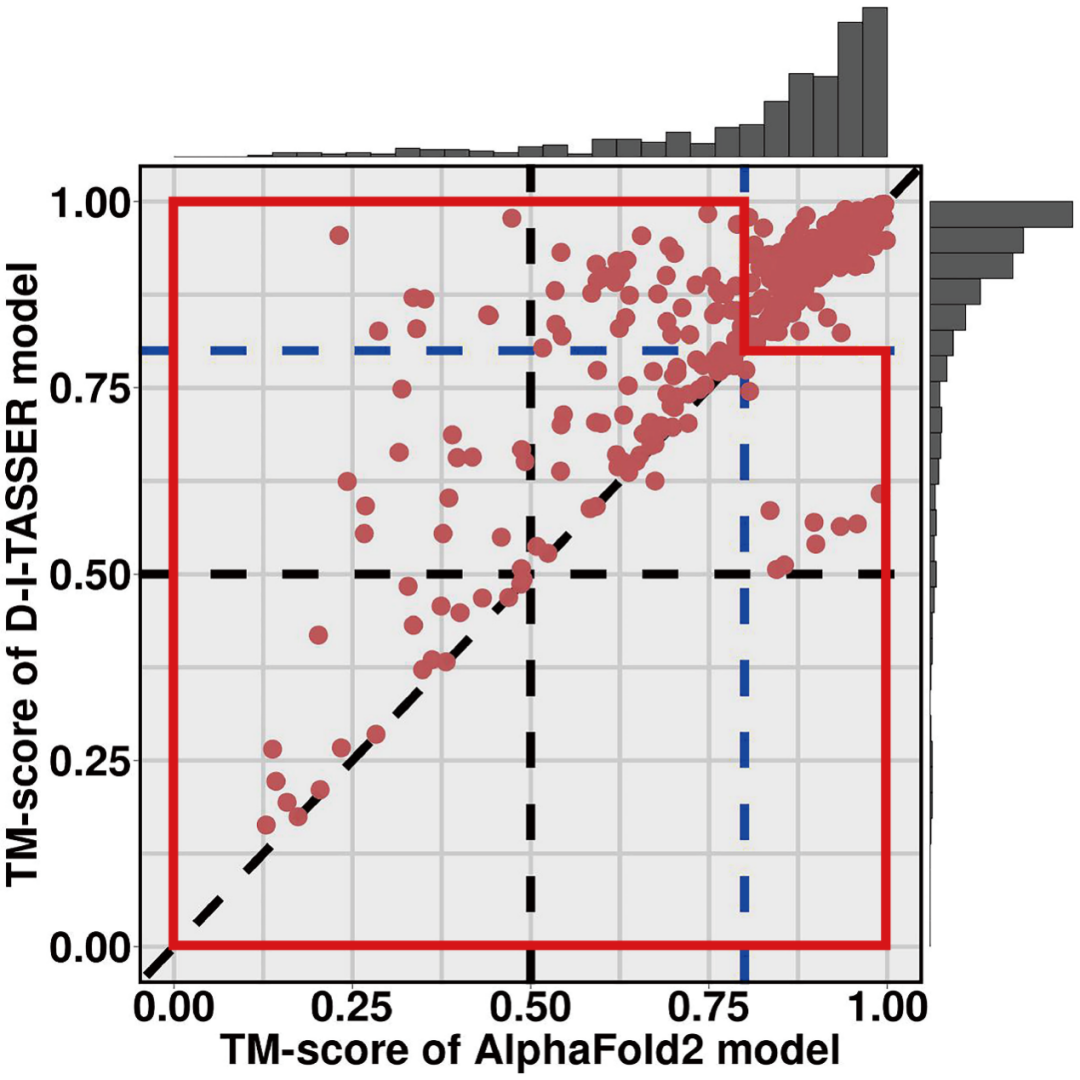 图4 D-I-TASSER与AlphaFold2.3在单结构域蛋白质上的性能比较 图4 D-I-TASSER与AlphaFold2.3在单结构域蛋白质上的性能比较
D-I-TASSER的优异表现得益于其整合了多种特征,包括深度多序列比对、多来源的深度学习空间约束预测器、融合深度学习及物理势的分子动态模拟等。图5对这些特征的贡献进行了系统性分析,随着不同类型约束的逐步融合,预测模型的TM-score持续提升。可见,D-I-TASSER预测精度的提升主要得益于深度学习获得的高精度空间约束。而在引入物理势之后,D-I-TASSER模型的TM-score达到0.870,超越AlphaFold2.3(0.829)和AlphaFold3(0.849),充分体现出深度学习与物理模拟协同驱动的强大建模能力,这种融合的模式要显著优于单一依赖深度学习、或者传统物理势的方法。
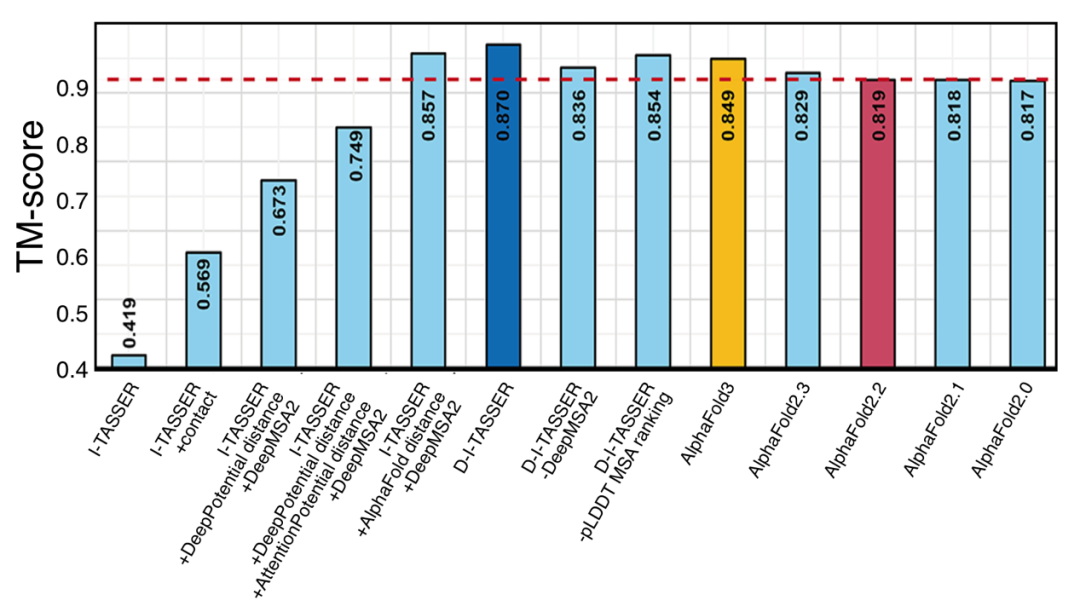 图5 D-I-TASSER与AlphaFold全系列、不同约束条件下的I-TASSER体系的比较 图5 D-I-TASSER与AlphaFold全系列、不同约束条件下的I-TASSER体系的比较
值得强调的是,深度学习空间约束的精度很大程度依赖多重序列比对,这是因为现代深度学习空间约束的一个主要训练特征正是多重序列比对中的共进化信息。高质量多序列比对的构建对D-I-TASSER的精度,起到了至关重要的作用。消融实验表明,移除DeepMSA2模块后,模型平均TM-score从0.870降至0.836(p-value=3.63E-69)。DeepMSA2通过丰富的宏基因组数据库和深度学习驱动的MSA筛选算法,显著提升了预测效果。即使将DeepMSA2的MSA直接用于AlphaFold2,也能小幅提高预测准确率(0.819→0.841),但整体性能仍不及D-I-TASSER(0.870)。这进一步验证,D-I-TASSER的卓越性能并非单一模块驱动,而是多源深度学习空间约束与物理势有效融合的结果。
为了直观揭示DeepMSA2与多源深度学习空间约束的协同效应,文章以来自番茄丛枝矮化病毒的RNA沉默抑制因子p19(PDB ID:4jgnA)为例(图6),深入剖析了不同模块对预测质量的影响。由于该蛋白质缺乏足够的同源序列,AlphaFold2构建的MSA较浅(Neff=0.36),因此其预测的结构质量较差(TM-score=0.335;图6A),其预测的距离图也存在较高误差(MAEn=3.20Å;图6C)。引入DeepMSA2后,MSA的比对深度提升了6.75倍,显著改善了AlphaFold2预测的距离图精度(MAEn=0.69Å;图6D)。然而,AlphaFold2的距离图仍然缺乏N端与其他区域之间的距离信息。D-I-TASSER进一步结合DeepPotential和AttentionPotential模型后,预测的距离图覆盖了整个序列区域,整体准确性进一步提高(MAEn=0.45Å;图6E)。依托这一复合距离图,D-I-TASSER最终获得了高质量预测结果(TM-score=0.871;图6B)。该案例表明,DeepMSA2构建的高质量MSA有效增强了进化特征的提取,而多源深度学习空间约束则进一步提升了空间信息预测的准确性与预测质量,两者的协同作用,是D-I-TASSER优异预测性能的关键所在。
注:MAEn是深度学习模型预测距离与实验结构的相应距离之间的平均绝对距离误差。
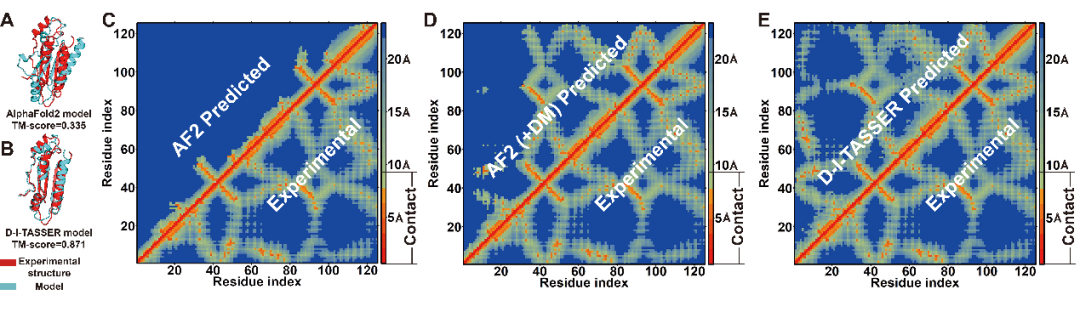 图6 番茄丛枝矮化病毒RNA沉默抑制因子p19上AlphaFold2与D-I-TASSER的预测结果比较。(A)AlphaFold2预测模型与实验比较;(B)D-I-TASSER模型与实验比较;(C)AlphaFold2接触图与实验比较;(D)AlphaFold2引入DeepMSA2之后的接触图与实验比较;(E)D-I-TASSER接触图与实验比较 图6 番茄丛枝矮化病毒RNA沉默抑制因子p19上AlphaFold2与D-I-TASSER的预测结果比较。(A)AlphaFold2预测模型与实验比较;(B)D-I-TASSER模型与实验比较;(C)AlphaFold2接触图与实验比较;(D)AlphaFold2引入DeepMSA2之后的接触图与实验比较;(E)D-I-TASSER接触图与实验比较
除了高质量的多重序列比对及多来源的深度学习空间约束外,D-I-TASSER的高精度预测更依赖于融合深度学习空间约束和物理势在折叠过程中的协同引导。以磷酸合酶(PDB ID:3fpiA)预测过程中的构象折叠轨迹为例(图7),在REMC模拟的前40轮分子模拟中,受两者的共同作用,预测结构的质量迅速提升:MAEm从7.7Å快速下降至1.2Å,同时TM-score从0.31跃升至0.71。随着模拟的进一步推进,预测构象在第100次轮时达到收敛状态,MAEm和TM-score分别稳定在0.39Å和0.96的高精度水平。这一轨迹分析表明深度学习空间约束与物理势的结合能够有效揭示蛋白质的折叠过程。
与以AlphaFold为代表的纯深度学习方法不同,图7所示的构象折叠轨迹展示了D-I-TASSER在重建蛋白质折叠路径方面的独特能力。长期以来,蛋白质在细胞环境中如何从无序链条折叠成有序三维结构的动力学过程,一直是生物物理学的重要研究课题。尽管深度学习方法在预测蛋白质最终稳定构型方面已取得显著突破,但其普遍缺乏对折叠动力学路径的建模能力,这一局限性也引发了科学界的广泛关注。D-I-TASSER通过将物理建模与深度学习相结合,不仅提升了结构预测精度,同时也为解析蛋白质折叠过程提供了一种新颖且可追踪的模拟框架,展现出在蛋白质动力学研究中的潜在应用价值。
注:MAEm是深度学习模型预测距离与D-I-TASSER模型相应距离之间的平均绝对距离误差。
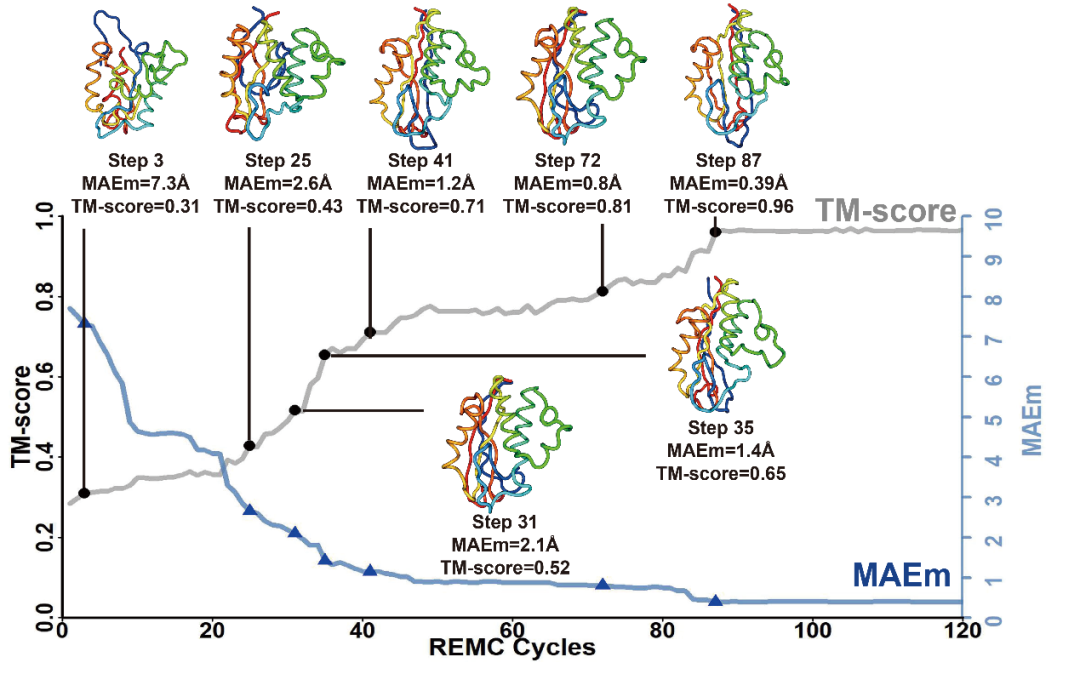 图7 D-I-TASSER在磷酸合酶(PDB ID: 3fpiA)结构预测过程中的构象折叠轨迹。 图7 D-I-TASSER在磷酸合酶(PDB ID: 3fpiA)结构预测过程中的构象折叠轨迹。
(二)多结构域蛋白质结构预测能力评估
D-I-TASSER在更复杂的多结构域蛋白质结构预测中整体优于AlphaFold2。为了系统比较D-I-TASSER与AlphaFold2的预测性能,文章构建了一个包含230个多结构域蛋白质(共557个结构域)的基准测试集。图8A和8B展示了D-I-TASSER与AlphaFold2在全链级别和结构域级别的结构预测性能比较。结果表明,D-I-TASSER预测的全链级别和结构域级别模型的TM-score分别为0.720和0.858,较AlphaFold2相应模型的TM-score(0.638和0.835)分别提高了12.9%和2.8%。进一步分析表明,该性能优势主要体现在难度较高的预测目标(图8A和8B所示的红色标注区域)上。在这些困难目标上,D-I-TASSER相对于AlphaFold2的TM-score提升分别为17.1%(全链级别)和9.9%(结构域级别),凸显了其在复杂结构预测中的显著优势。
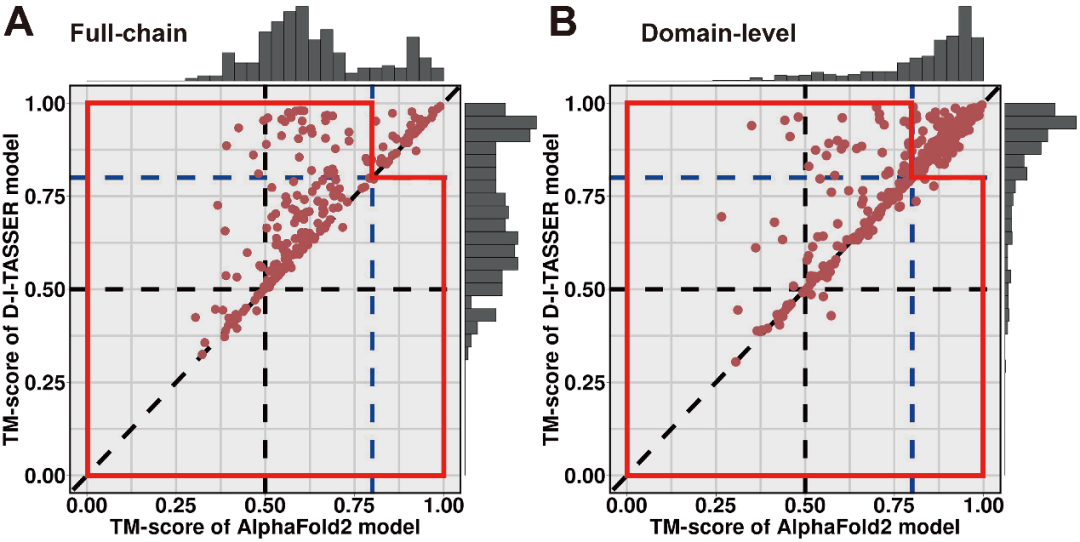
图8 D-I-TASSER与AlphaFold2在多结构域蛋白质上的性能比较
(A:全链;B:结构域)
定量的结果分析表明,D-I-TASSER主要通过两个方面来提升多结构域蛋白质结构预测的质量:(1)通过结构域划分策略,提升结构域级别预测结构的质量;(2)通过结构域特征组装策略,提升结构域间空间约束质量及全链级别预测结构的质量。
针对D-I-TASSER提升结构域级别预测结构质量方面,文章分析了C. reinhardtii鞭毛辐射辐条蛋白质(PDB ID:7jtkB)这一双结构域蛋白质的预测案例(图9)。由于检测到的有效序列数量较少(Neff=0.1),AlphaFold2预测的全链模型质量较差(全链TM-score=0.425),且结构域内的预测效果也不佳。相比之下,D-I-TASSER通过结构域划分与组装模块,提高了MSA的覆盖度(Neff=0.4),同时分别为两个结构域检测到额外的688条和15条同源序列,显著改善了距离图预测准确性。在更准确的约束引导下,D-I-TASSER最终预测了高质量的结构模型(全链TM-score=0.934),两个结构域的TM-score分别为0.971和0.910,远优于AlphaFold2的结果。该案例表明,在有效同源序列稀缺的情况下,结构域划分与组装的MSA构建策略,能够显著提升结构域内的MSA质量及预测结构质量,进而提升全链模型预测准确性,这是D-I-TASSER能够克服多结构域挑战的关键因素之一。
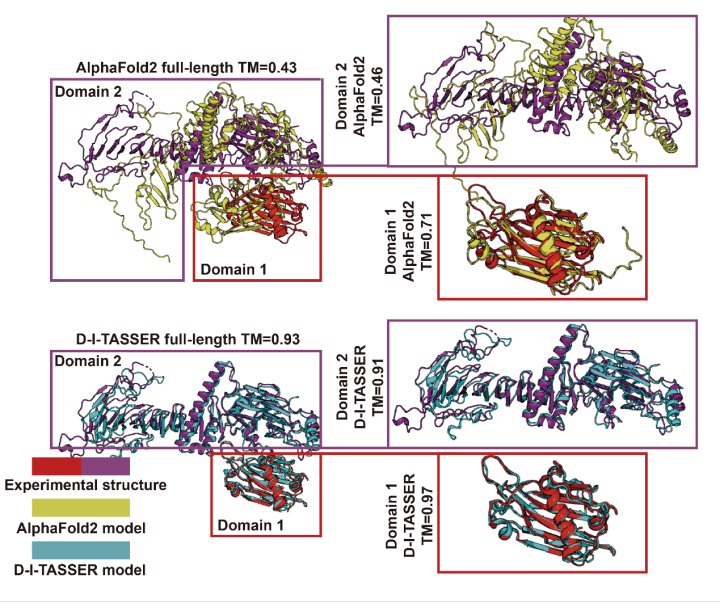 图9 C. reinhardtii鞭毛辐射辐条蛋白质上AlphaFold2与D-I-TASSER的预测结果比较 图9 C. reinhardtii鞭毛辐射辐条蛋白质上AlphaFold2与D-I-TASSER的预测结果比较
针对D-I-TASSER提升结构域间空间约束质量方面,文章考察了InaD-like蛋白质(Inactivation No Afterpotential D,PDB ID:6irdC)这一双结构域蛋白质。虽然 AlphaFold2能较为准确地预测各结构域的局部构象(TM-score分别为0.894和 0.930),但由于多序列比对信息有限,其预测结构域间的空间排布严重错误,导致整体模型偏离真实构象,全链TM-score仅为0.510(图10上图)。相比之下,D-I-TASSER在划分结构域后,借助比对深度更深的MSA,通过全链和结构域的MSA组装,有效提升了预测精度(全链TM-score达0.890,图10下图)。该结果表明,引入的结构域特征组装模块可有效挖掘更丰富的进化信息,从而提升多结构域蛋白质结构域间的预测质量,进而提升结构预测的准确性,这是D-I-TASSER能够成功预测多结构域蛋白质结构的重要原因。
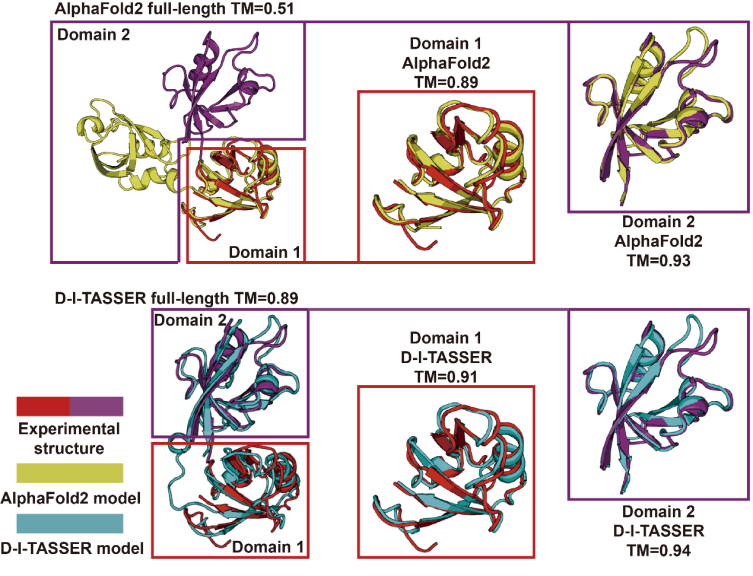 图10 人类InaD-like蛋白质上AlphaFold2与D-I-TASSER的预测结果比 图10 人类InaD-like蛋白质上AlphaFold2与D-I-TASSER的预测结果比
文章发现,D-I-TASSER融合了深度学习空间约束与物理势,通过REMC方法模拟生成了大量多样化的构象(decoys),为预测多构象状态的蛋白质提供了巨大潜力。图11展示了一个典型案例——新冠病毒(SARS-CoV-2)刺突蛋白质三聚体,其蛋白质链可以处于开放(open)或闭合(closed)两种构象状态(图11A)。这两种状态的主要差异来源于,与C端受体结合的结构域(RBD)相对于其他结构域发生了方向变化。D-I-TASSER成功预测了这两种构象(图11B),其中,第一个模型对应闭合状态(TM-score=0.94),第二个模型对应开放状态(TM-score=0.99)。如图11C所示,D-I-TASSER生成的构象大致可以分为开放、闭合和中间状态三类。SPICKER聚类分析显示,这些构象可以被聚类为五个簇,第一个模型(闭合状态)和第二个模型(开放状态)分别来自其中最大和次大的簇。相比于通常只生成单一静态模型的纯深度学习方法,D-I-TASSER借助物理模拟能够全面探索构象空间,并通过聚类有效识别出多种构象状态。图11所示结果凸显了D-I-TASSER基于物理的模拟在捕捉蛋白质多构象状态方面的内在优势。
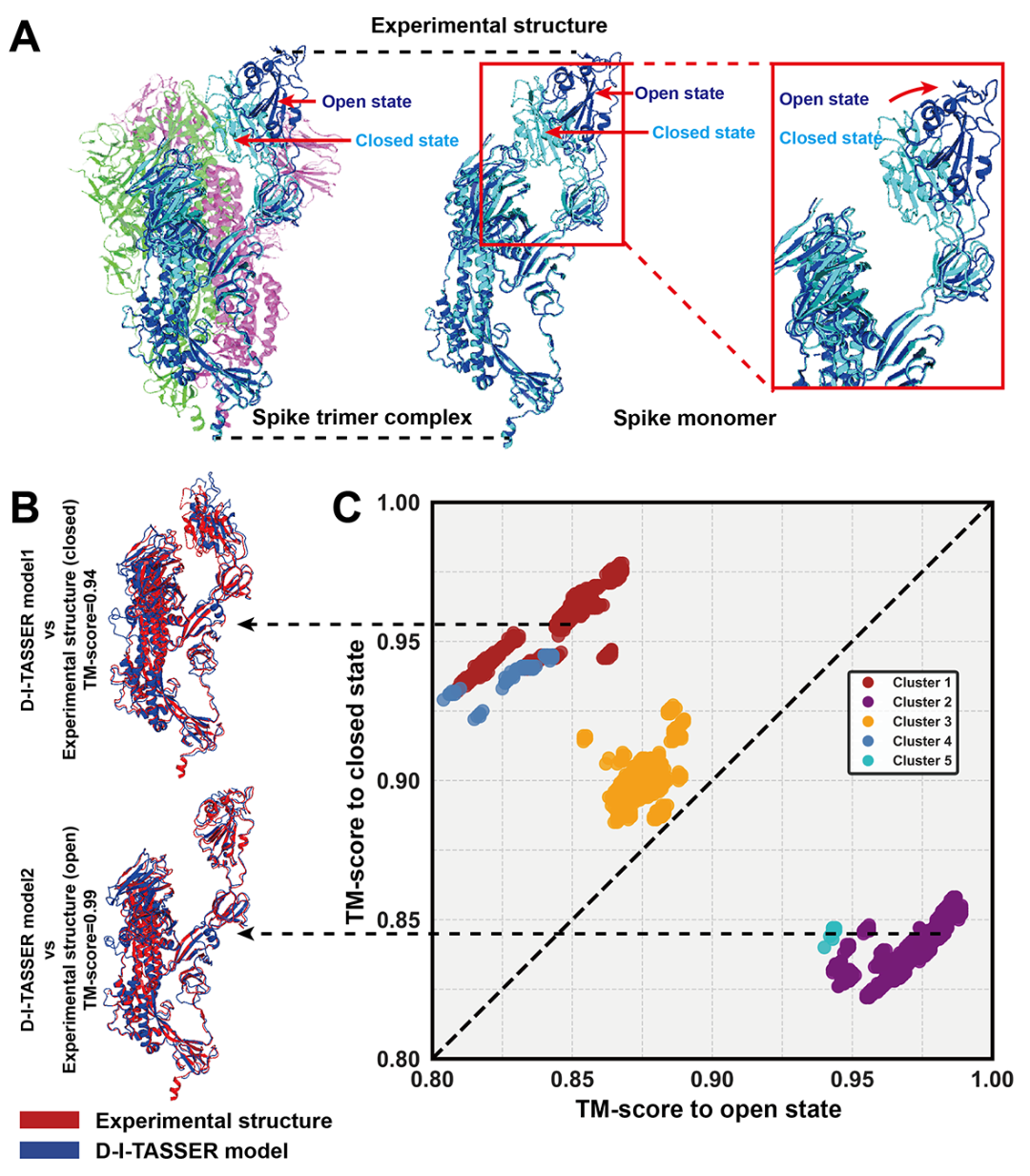 图11 D-I-TASSER成功预测出新冠病毒(SARS-CoV-2)刺突蛋白质的开放和闭合两种构象状态 图11 D-I-TASSER成功预测出新冠病毒(SARS-CoV-2)刺突蛋白质的开放和闭合两种构象状态
D-I-TASSER算法(参赛名:UB-TBM)参加了第15届世界蛋白质结构预测大赛(CASP15),并在单结构域预测(Regular)和多结构域预测(Inter-domain)比赛中均排名第一(图12A和12B)。CASP是国际公认的,世界范围蛋白质结构预测权威竞赛,每两年举行一次,旨在通过对大分子和复合物结构预测计算方法进行严格的评估,来推动结构预测领域技术的进步。为了保证结果的客观性,CASP采用严格的双盲预测机制,长期以来被视为结构预测领域的“金标准”,并被业界誉为“蛋白质结构预测的奥林匹克”。郑伟博士从2016年开始参加CASP比赛并取得优异成绩,累计获得十项单项冠军。张阳教授团队开发的计算机算法自2006年以来连续9次获得CASP大赛冠军。
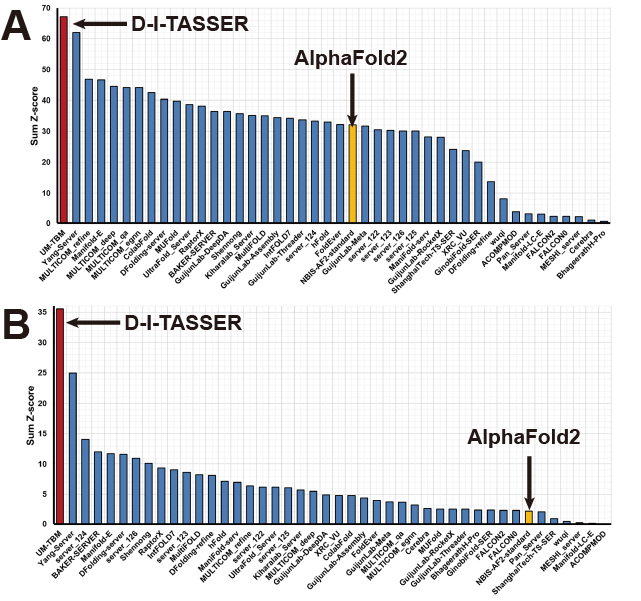 图12 CASP15世界蛋白质结构预测大赛单结构域(A)和多结构域(B)预测比赛排名 图12 CASP15世界蛋白质结构预测大赛单结构域(A)和多结构域(B)预测比赛排名
按照CASP15组委会公布的官方Z-score评分结果,D-I-TASSER的整体表现优于其他所有参赛组。在单结构域和多结构域目标中,D-I-TASSER的累计Z-score分别为67.20和35.53,显著高于AlphaFold2的32.05和2.11。值得一提的是,CASP15包含两个赛道:服务器赛道要求预测模型在72小时内提交;而人工赛道允许专家手动干预,并为每个目标预留最长三周的预测时间。即便在与人类专家组同场竞争的情况下,D-I-TASSER自动服务器在单结构域预测目标中依然取得了第二名(若按Z-score>0.0统计则为第一名)的优异成绩。此外,在多结构域预测中,D-I-TASSER服务器的表现明显优于包括人类专家组在内的其他所有参赛组,其累计Z-score比排名第二的参赛组(24.96)高出42.3%。
作为蛋白质结构预测领域的里程碑式方法,AlphaFold系列方法凭借其革命性的端到端深度学习框架,持续引领着单链结构预测的技术革新。然而在复杂多结构域及困难折叠单元的预测任务中,融合了深度学习与物理建模的D-I-TASSER方法展现出更强的预测能力——如图13所示,通过对困难(FM)蛋白质结构域(50个)和多结构域蛋白质(20个)的TM-score对比分析,D-I-TASSER预测结果均显著优于包括AlphaFold3在内的所有AlphaFold系列方法。
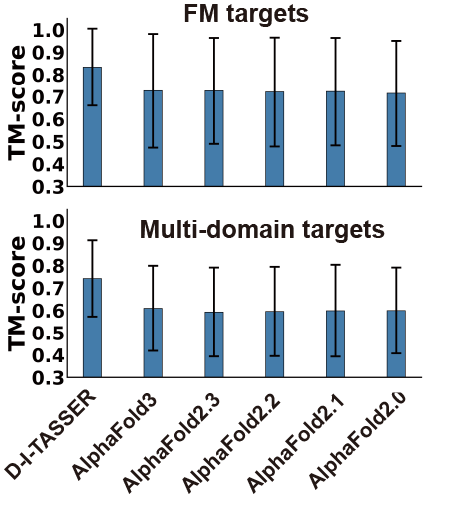 图13 CASP15中困难(FM)蛋白质结构域和多结构域蛋白质上D-I-TASSER与AlphaFold全系列的TM-score比较 图13 CASP15中困难(FM)蛋白质结构域和多结构域蛋白质上D-I-TASSER与AlphaFold全系列的TM-score比较
文章选取了19个单结构域及8个多结构域目标作为代表性案例,其中D-I-TASSER相较于AlphaFold2的TM-score提升均超过0.15分差(图14)。值得关注的是,D-I-TASSER成功预测了多个大尺寸(残基数大于3,000)多结构域蛋白质的三维结构,例如T1169(残基数3,364)预测模型的TM-score达到0.8。这一系列成果标志着深度学习约束在大尺寸蛋白质结构预测领域取得了重要进展,突破了传统方法长期以来所面临的长链蛋白结构预测的挑战。
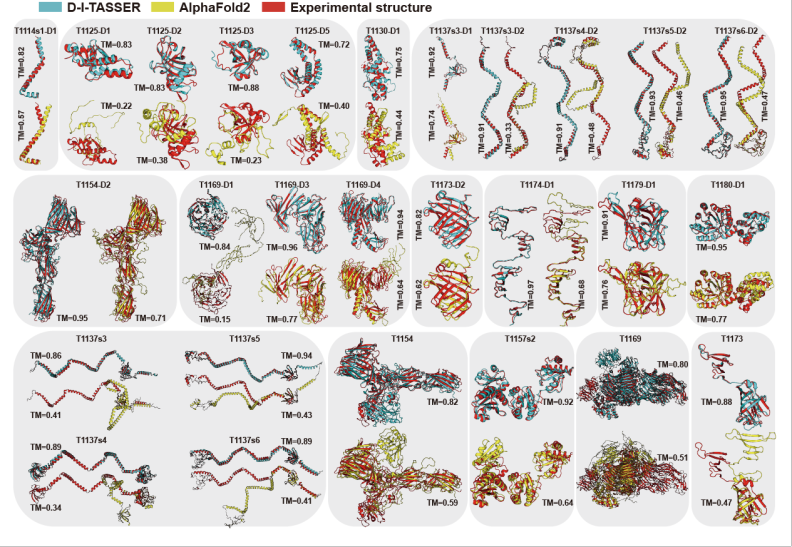 图14 CASP15中D-I-TASSER与AlphaFold2在部分靶标上的预测结果比较(绿色和橙色分别代表D-I-TASSER和AlphaFold2的预测模型,红色为实验解析结构)。 图14 CASP15中D-I-TASSER与AlphaFold2在部分靶标上的预测结果比较(绿色和橙色分别代表D-I-TASSER和AlphaFold2的预测模型,红色为实验解析结构)。
最后,D-I-TASSER对人类基因组的19,512个蛋白质和34,968个结构域进行了结构预测,涵盖了约95%的人类蛋白质组。为评估模型质量,D-I-TASSER设计了eTM-score作为预测精度的估计指标。结果显示,D-I-TASSER成功预测了80.5%的单结构域和72.8%的全链蛋白质结构(图15A),且预测结果与AlphaFold2数据库高度互补。值得注意的是,D-I-TASSER还准确预测了3,020个AlphaFold2难以可靠预测的人类蛋白质,凸显其在填补结构预测盲区方面的重要价值。进一步分析表明,预测结构的eTM-score在各染色体间分布均匀(图15B),证实预测精度与基因在染色体上的定位无显著相关性。此外,D-I-TASSER还对人类蛋白质组的生物学功能进行了系统注释(图15C),包括基因本体功能标签(GO)、酶分类(EC)以及小分子绑定位点(LBS)。
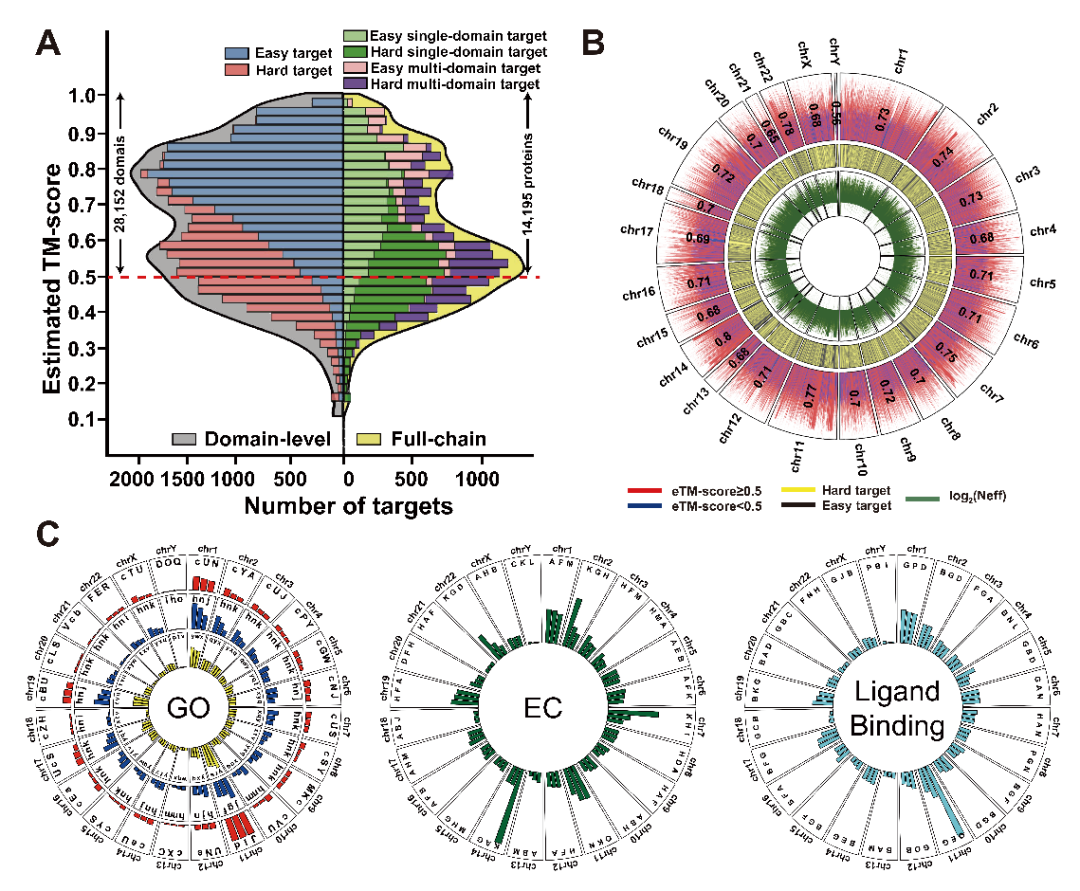 图15 (A) D-I-TASSER在人类蛋白质组上的eTM-score分布;(B) 对eTM-score分布(外道)、目标类型(中道)和Neff对数(内道)的染色体级别分析;(C) 基于D-I-TASSER的人类蛋白质组功能注释。 图15 (A) D-I-TASSER在人类蛋白质组上的eTM-score分布;(B) 对eTM-score分布(外道)、目标类型(中道)和Neff对数(内道)的染色体级别分析;(C) 基于D-I-TASSER的人类蛋白质组功能注释。
本研究在I-TASSER和深度学习的基础上,开发了一个新的蛋白质结构预测算法D-I-TASSER。该方法通过整合多源深度学习空间约束及物理势,并引入全新的结构域划分与组装模块,实现了大尺寸单结构域及多结构域蛋白质的自动化预测。大规模基准测试结果表明,D-I-TASSER在单结构域与多结构域蛋白质预测方面均显著优于主流的AlphaFold系列方法。D-I-TASSER的卓越性能得益于多种深度学习空间约束及物理势的有效融合。新引入的结构域划分与组装模块可有效挖掘更丰富的进化信息,从而提升多结构域蛋白质预测的准确性。特别是,在国际权威的CASP15盲测中,D-I-TASSER在单结构域和多结构域预测两个任务类别中均取得了最佳表现,验证了其在实际应用中的广泛适用性和领先水平。
总体而言,D-I-TASSER的成功展示了将深度学习与物理模拟有机结合的巨大潜力,不仅在复杂蛋白质和多结构域蛋白质的结构预测中取得了显著进展,也为未来的研究提供了清晰的路线图:通过深度学习赋能物理模拟,推动蛋白质结构预测迈向更高精度和更广适用性的阶段。另外,D-I-TASSER算法框架具有良好的可扩展性,能够不断整合更高精度的深度学习方法,进一步应对孤儿蛋白质或蛋白质复合物结构预测问题中的挑战。
值得一提的是,当前蛋白质结构预测方法仅部分解决了蛋白质折叠领域的核心问题。这一挑战主要体现在两个方面:首先,蛋白质在细胞环境中并非保持静态构象,而是持续经历热力学涨落。这种动态特性对蛋白质的生物学功能至关重要,尤其是在药物设计领域,因为蛋白质-药物相互作用往往依赖于动态构象之间的平衡。然而,现有的深度学习方法(如AlphaFold)通常仅能预测单一静态结构,难以捕捉这种构象动态性。其次,从基础科学的角度,理解蛋白质如何从线性氨基酸序列折叠成复杂三维结构(即折叠路径问题)仍是未解之谜。尽管AI在高精度结构预测方面取得突破,但其对折叠动态过程的建模能力仍然有限。在这一背景下,D-I-TASSER通过融合深度学习与物理模拟方法,展现出较为独特的优势。一方面,它能够生成大量可行的构象集合,通过聚类分析可揭示蛋白质在类细胞环境中的构象涨落行为与可能存在的可替代折叠态(alternative states);另一方面,其基于动态模拟的建模框架,可在一定程度上追踪蛋白质从无序链条逐步折叠至稳态结构的路径,为构建蛋白质折叠动力学模型提供了新手段。尽管上述构象涨落和折叠路径的预测结果仍需进一步通过实验手段进行严格验证,但这一框架所具备的内在物理一致性和构象可解释性,为解决传统AI方法在蛋白质折叠研究中的关键瓶颈提供了新的思路。
从技术交叉与应用拓展角度看,该成果还具备潜在的延伸价值:在药物设计领域,该成果对蛋白质结构有着高效的解析能力,如果能结合相干光量子计算机量子并行性加速分子动力学模拟的优势结合,或可进一步缩短新药研发周期。从长远看,待量子机器学习技术成熟,D-I-TASSER 的深度学习模块或可迁移至量子计算框架,借助量子特性优化蛋白质折叠路径模拟,不过这些关联性仍需跨学科研究进一步验证。
注:D-I-TASSER服务器https://zhanggroup.org/D-I-TASSER/,对科研人员免费开放。
作者简介
郑伟(论文第一作者),南开大学统计与数据科学学院教授,博士生导师。长期从事基于深度学习及物理势的生物分子及其互作的结构预测研究,主持开发C-I-TASSER、C-QUARK、D-I-TASSER、DMFold等结构预测算法,累计获得CASP国际大赛十项冠军,多次受邀在国际会议做特邀报告。郑伟博士在Nature Biotechnology、Nature Methods、Nature Communications、PNAS等期刊发表文章50余篇,其主导开发的结构预测算法服务于100个国家的近10万名用户。
乌云其其格(论文共同第一作者),密歇根州立大学计算机科学与工程系博士,于南开大学获得生物信息学硕士学位和信息与计算科学学士学位。研究方向涵盖计算生物学、生物信息学、系统进化、蛋白质结构预测及其在疾病机制与药物研发中的应用。研究成果在Nature Biotechnology、Nature Methods、Nucleic Acids Research等多种国际期刊及会议发表。
李阳(论文共同第一作者),新加坡国立大学癌症科学研究所特别研究员及资深研究科学家,AI和深度学习专家,专注于深度学习在结构生物信息学和生物医学中的应用与研究。
Lydia Freddolino(共同通讯作者),密歇根大学生物化学系副教授。
张阳(通讯作者),新加坡国立大学计算机科学和生物化学教授,新加坡癌症科学研究院高级首席研究员,常年从事基于人工智能的蛋白质和RNA结构预测及药物设计研究,其团队开发的计算机算法连续9次获得国际CASP大赛冠军。
| 






















