计算蛋白质设计旨在生成具有特定结构、功能或动态特性的蛋白质序列与三维结构。当前蛋白质设计主要依赖生成模型(如扩散模型、幻觉方法)从分布中采样,流程多为先生成主链结构再设计序列。尽管扩散模型在小蛋白设计中表现出色,但其O(N³)复杂度限制了在长序列上的应用,且针对新任务需重新训练或定制能量函数,适应性较差。
为了突破现有挑战,7月的nature machine intelligence上提出了SALAD——一组稀疏全原子去噪模型,它能够更快、更高效地生成长达1000个残基的蛋白质结构,且设计质量相当或更优;同时结合结构编辑策略,无需重新训练即可灵活应用于基序支架设计、多态设计等多种复杂任务。

稀疏蛋白质模型架构
模型架构基于稀疏等变变换器(图1a),通过限制每个残基的注意力计算至其局部邻居(图1b),将复杂度从O(N²)降至O(N·K)。该架构采用IPA层和GeGLU前馈层,直接更新原子位置并动态计算框架以保持等变性。在PDB数据上训练的自编码器模型(图1c)验证了其有效性:所有变体在CASP14/15测试集上经少量迭代即可实现<1Å的重建精度(图1d),最终选定无需距离图邻居的等变版本作为基础架构。
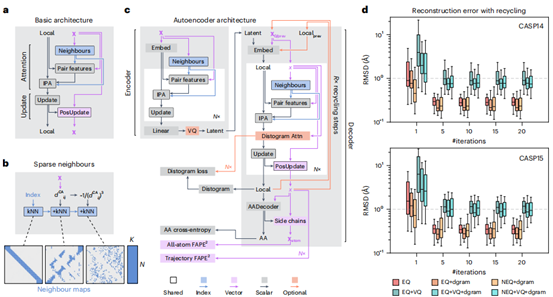
图1 | 稀疏模型架构
编辑去噪蛋白质模型
SALAD模型通过训练去噪扩散模型来生成包含主链和侧链原子的全原子结构(图2a),其推理过程通过迭代去噪实现(图2b)。模型引入了可定制的编辑函数以适配不同设计任务。运行时间基准测试显示(图2c),SALAD在生成速度上显著优于现有先进模型(最快达7倍),尤其在长序列生成上优势巨大(1000残基仅需19秒),实现了高效、参数更少的蛋白质生成。
稀疏模型生成多样化且可设计的蛋白质结构
SALAD模型在主链生成质量评估中表现出色(图3a-c),其VP模型在50-400残基范围内与先进模型相当,而VP缩放和VE模型能有效生成长达1000残基的可设计主链(设计能力最高达36.7%)。通过形状噪声初始化(图3d),模型可生成特定形状(如字母)的高质量主链。在多样性方面(图3e,f),其可设计结构的多样性与RFdiffusion相当,并在大蛋白上超越Genie 2。
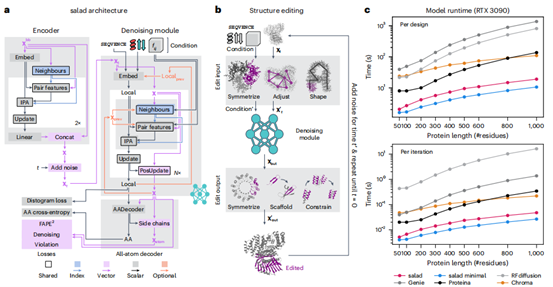
图2 | 去噪模型架构和运行时间
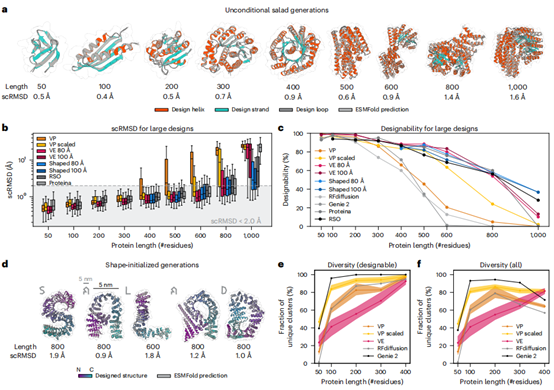
图3 | 使用salad模型进行无条件结构生成
随机二级结构条件化最大化多样性
通过随机二级结构条件化生成蛋白质主链(图4a),显著提升了生成结构的多样性(图4b)并平衡了二级结构含量偏差(图4c)。基于50,000个合成主链的分析表明,81.4%结构可设计,其中91.4%可聚类为多样折叠(图4d-e),且75.3%代表结构可设计,成功探索了PDB之外的“暗物质”折叠空间。
合成数据提高了一次性设计能力
在合成数据集上训练的蛋白质生成模型(图4f)显著优于基于PDB训练的模型:对于50-256残基的蛋白质,其生成的主链-序列对具有更低的中位scRMSD和更高设计成功率(>75%),而PDB训练模型设计成功率低于20%。该模型直接生成可折叠主链与序列,将序列设计步骤从8次减少至1次,大幅提升设计效率。
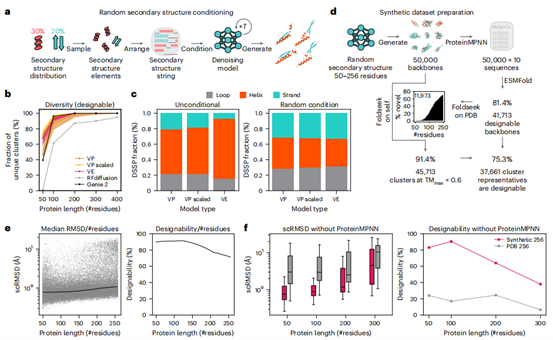
图4 | 随机二级结构条件化在牺牲设计能力的情况下最大化多样性
结构编辑用于基序支架设计
在基序支架设计基准测试中,salad模型通过两种策略(结构编辑salad+edit与多基序条件化salad+cond)与Genie 2、RFdiffusion对比。两种策略均成功解决24个单基序中的23个及6个多基序中的5个任务,其中salad+cond生成1,610个独特支架(略优于Genie 2),显著优于RFdiffusion(889个)。尽管各模型在不同任务中表现存在差异,但salad在多数任务中生成相同或更多支架,证明其在单/多基序支架设计中具备与最先进方法竞争的实力。
结构编辑用于重复蛋白设计
通过结构编辑结合对称化操作(图5a,c),作者的模型成功生成点对称(如循环对称)与螺旋对称的重复蛋白(图5b,d)。生成的主链具有低scRMSD和结构多样性(全α至全β拓扑),经ProteinMPNN设计后即使扩展3倍重复数仍能保持结构稳定性(图5e)。设计成功率受对称参数(如半径、旋转角)影响,紧凑结构(如小半径)会降低设计能力,证明该方法可灵活生成几何可控的重复蛋白。
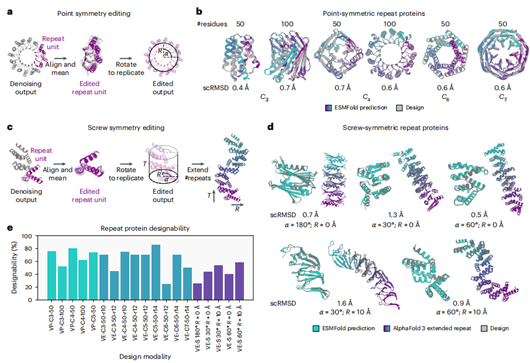
图5 | 对称和重复蛋白的编辑去噪
结构编辑用于多态设计
通过结构编辑耦合多个去噪过程(图6a),作者成功解决了多态蛋白设计任务:生成的主链-序列对在母体与子体状态间实现构象转换(β折叠→α螺旋)。其中2.9%的主链完全满足设计标准(AlphaFold RMSD<3.0 Å,pLDDT>75),成功率较ProteinGenerator提升6倍(图6b,d)。仅使用二级结构条件化时成功率降至0.4%,证明结构编辑是性能提升的关键。该工作首次实现了基于主链去噪模型的多态设计。
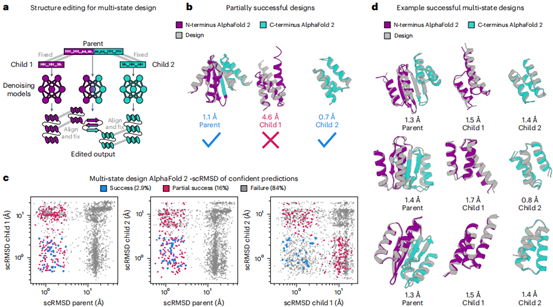
图6 | 多态蛋白质设计
总结
本研究提出的salad模型是一组高效的稀疏去噪模型,能够生成长达1000个残基的可设计蛋白质结构。其在设计能力与多样性上超越RFdiffusion,并与Genie 2性能接近,同时大幅降低计算成本。结合结构编辑技术,salad可灵活应用于基序支架、重复蛋白(包括新型螺旋对称结构)及多态设计等复杂任务,在多态基准任务中成功率提升一个数量级。虽然缺乏实验验证且训练数据限于PDB,但其基于ESMFold/AlphaFold的评估流程与已验证模型一致,证实了实用性。salad以高效、通用的特性推动了长链蛋白质设计的边界。
原文链接:https://www.nature.com/articles/s42256-025-01100-z
文章改编转载自微信公众号:北京生物结构前沿研究中心
原文链接:https://mp.weixin.qq.com/s/Q_NKTgmFhbBRPXSP2GUEyw | 


























