核磁共振(NMR)波谱学一直被视为有机分子结构解析的“金标准”,然而,其传统解析流程高度依赖专业人员的经验,不仅耗时费力,在面对复杂天然产物或低信噪比样品时更显得效率不足。尽管人工智能技术已被尝试用于辅助NMR解析,但现有方法多局限于单维谱图匹配或局部结构片段拼接,尚未实现从原始多维NMR数据到完整分子结构的端到端精准推断。
近期,中国医学科学院药物研究所汪小涧研究团队联合相关课题组,提出了一种多维NMR解析模型——NMRMind,实现了从1D/2D NMR谱图直接生成分子结构的端到端智能解析。相关成果以“NMRMind: A Transformer-Based Model Enabling the Elucidation from Multidimensional NMR to Structures”为题发表于国际化学权威期刊《Analytical Chemistry》。

NMRMind模型的核心创新
本研究研发了一种基于Transformer架构的深度学习模型——NMRMind,能够直接从多维NMR谱图(包括¹H、¹³C、COSY、HSQC、HMBC等)生成完整的分子SMILES结构(图1)。与传统计算机辅助结构解析(CASE)系统依赖规则或片段库不同,NMRMind通过大规模预训练与多模态微调,构建了从谱图信号到化学结构的“智能映射”。
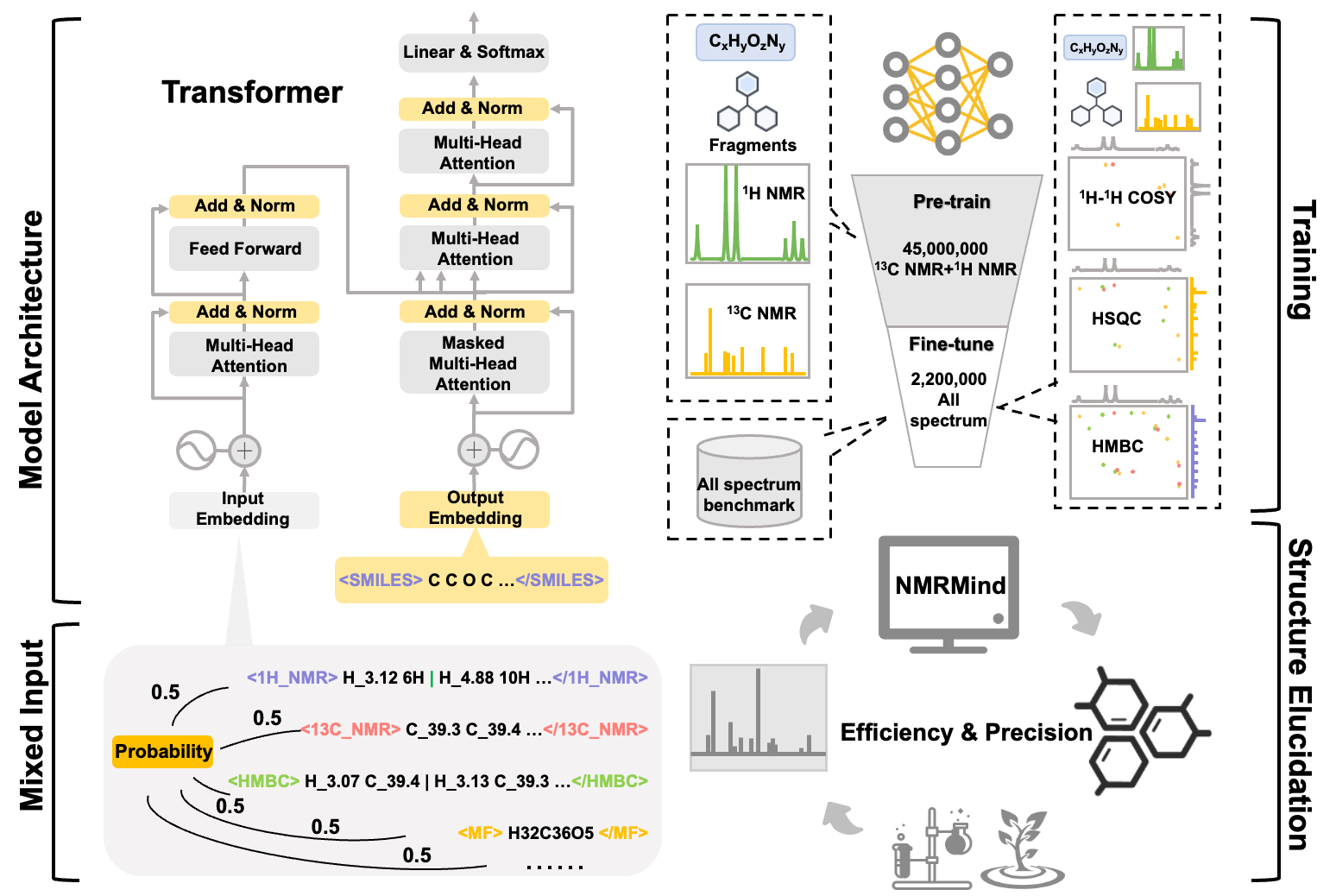
图1. NMRMind模型架构与工作流程示意图。多维NMR谱图经离散化编码后输入Transformer编码器,解码器输出SMILES结构。输入谱图表示方式:1D谱以化学位移token表示(如H_7.21),2D谱以坐标对形式(如C_120.5|H_6.89)输入。模型应用于天然产物结构解析和有机合成中未知产物的结构推断。
研究团队构建了迄今最大规模的1D&2D NMR-结构配对数据集,包含220万条分子及其模拟的1D/2D NMR谱图,并利用4500万条¹H/¹³C谱图进行预训练。其次,NMRMind引入混合模态输入机制,可灵活整合分子式、结构片段等先验信息,并采用混合模态dropout策略增强模型在谱图缺失情况下的鲁棒性。
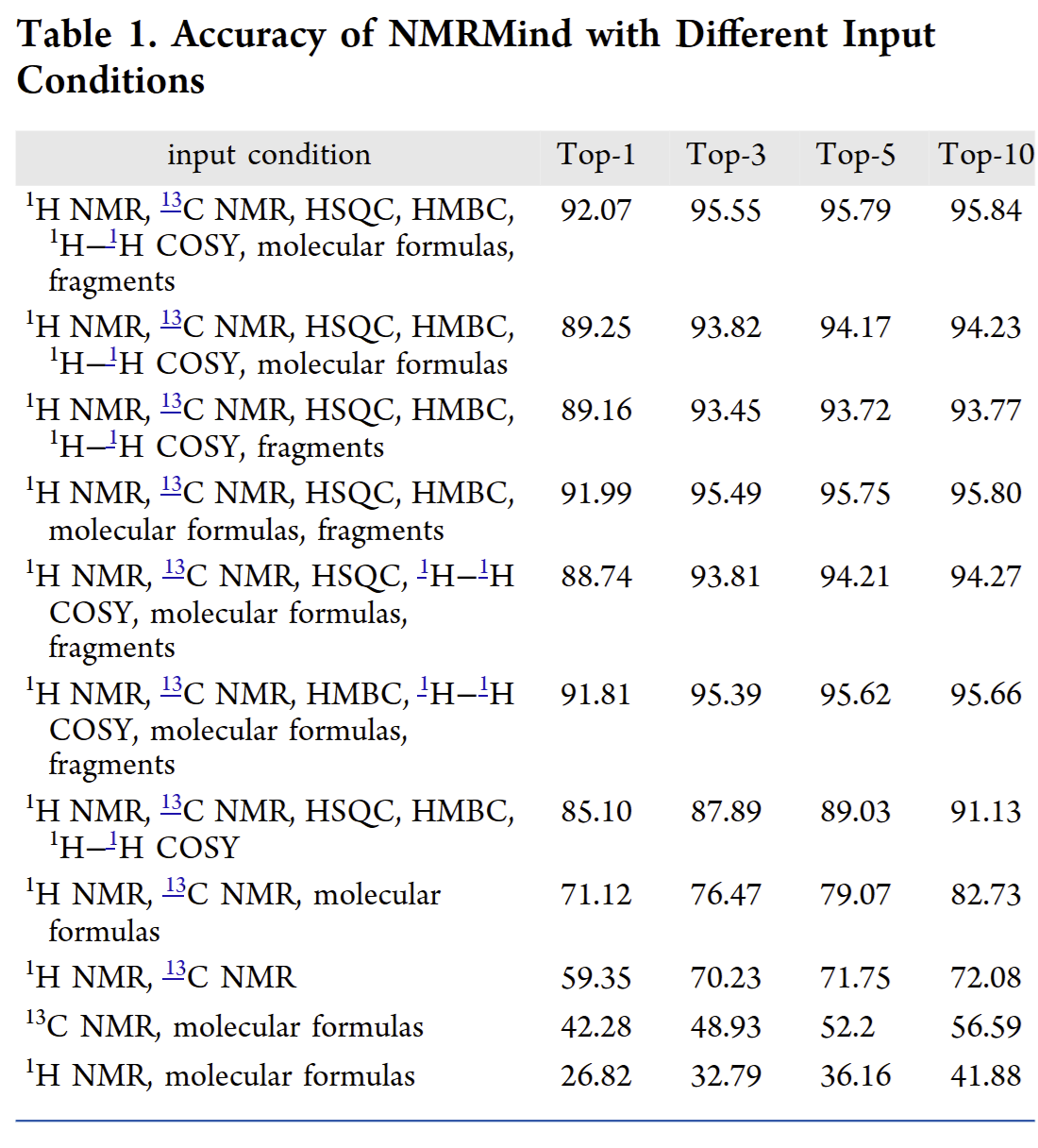
表1. NMRMind在不同输入条件下的准确性
卓越的性能与实际应用验证
研究表明,NMRMind在多种输入条件下均表现出极高的准确性。如表1所示,在输入完整多维NMR数据时,其Top-1结构准确率高达92.07%,且单次解析耗时不足0.05秒。即便仅使用NMR谱图(不提供分子式),其准确率仍保持在85.10%的高水平。
更为重要的是,NMRMind在真实场景中得到了成功验证。在天然产物研究中,该模型辅助鉴定了厚朴提取物中的6个全新化合物。
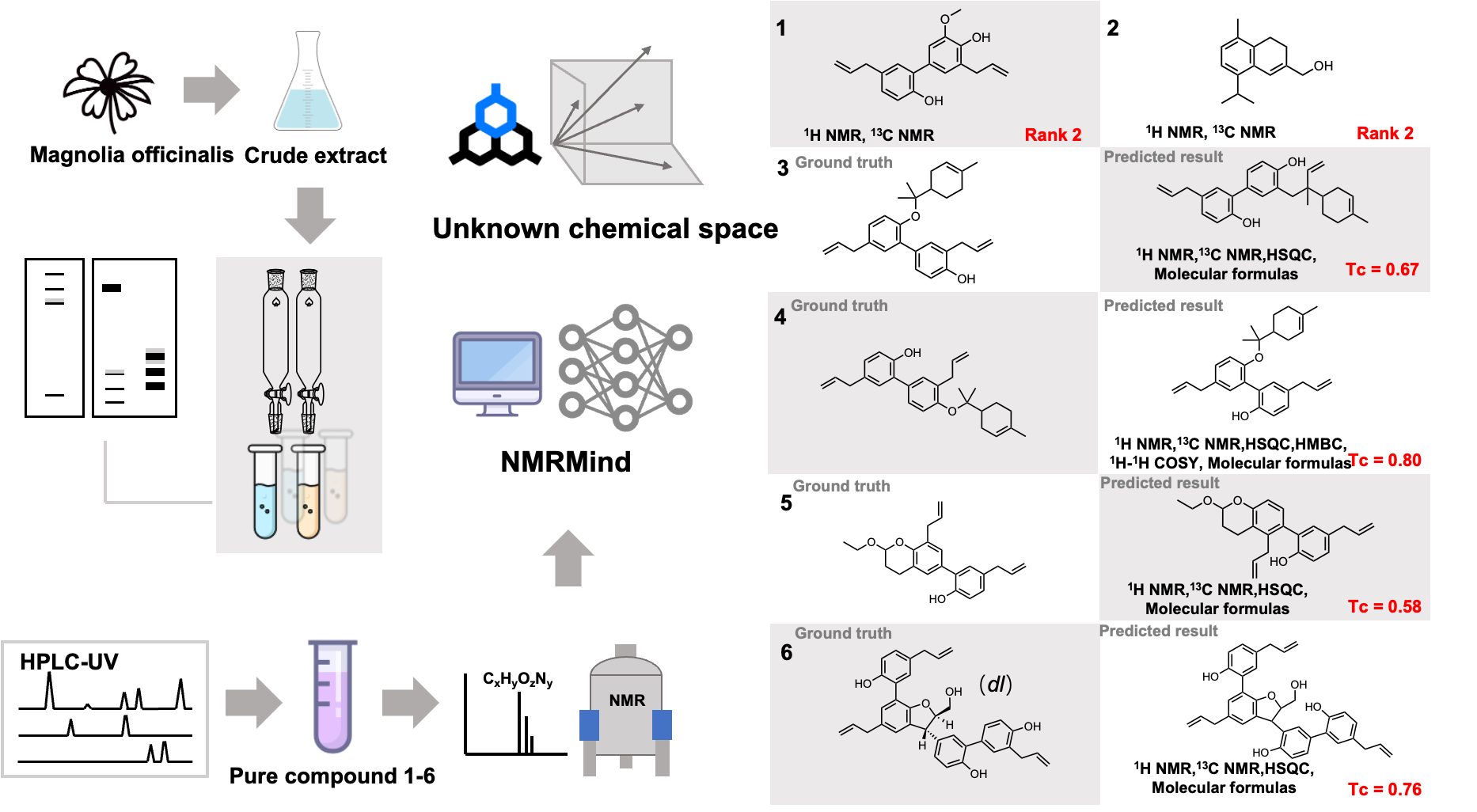
图2. 真实实验验证:厚朴提取物中6个新天然产物的AI辅助解析结果。NMRMind预测结构与最终经人工验证的正确结构对比,显示高一致性。化合物6的PTP1B抑制活性验证,证实其药物潜力。
其中,化合物6被发现具有强效的PTP1B抑制活性(IC₅₀ = 1.97 μM),显示出显著的药物开发潜力。此外,在有机合成领域,NMRMind被用于识别反应中难以预料的副产物结构。这为快速阐明反应机理提供了强大工具,显著加速了合成路线优化。
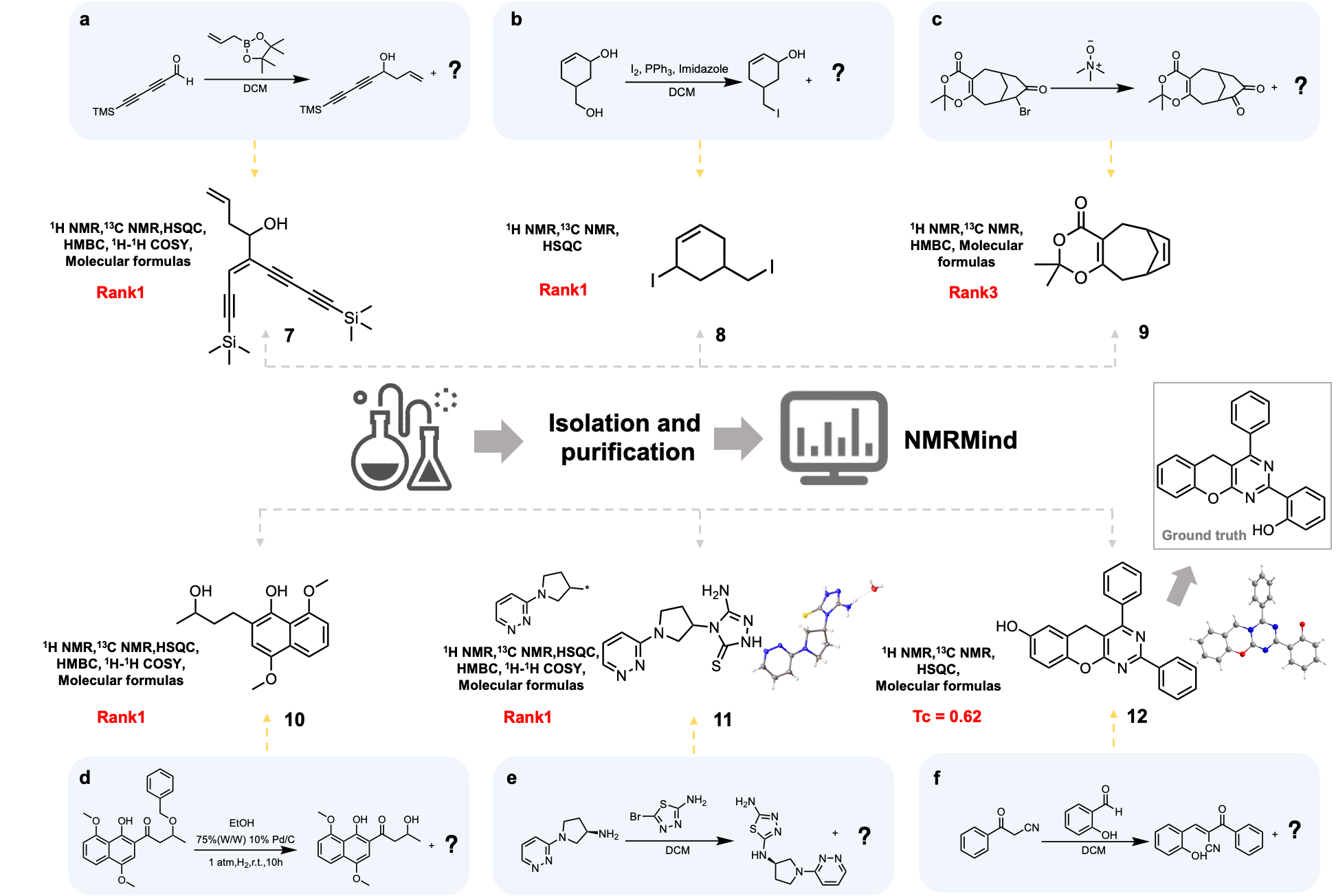
图3. NMRMind迅速识别出由六个有机反应(a-f)产生的意外产物。
总结与展望
本研究研发的NMRMind模型,成功突破了传统NMR结构解析的效率与精度瓶颈。其端到端的智能解析能力,不仅极大地缩短了分子结构鉴定周期,更在天然产物发现、反应机理解析及药物先导化合物筛选等领域展现出巨大的应用潜力。此项工作标志着化学结构表征正迈向“AI驱动”的新范式,并为全球化学智能社区的发展提供了重要的工具与数据基础。
参考资料
Xue, X., Sun, H., Sun, J., Patiny, L., Liu, X., Chen, K., Yan, J., Li, L., Liu, X., Xu, S. and Zhang, D., 2025. NMRMind: A Transformer-Based Model Enabling the Elucidation from Multidimensional NMR to Structures. Analytical Chemistry.
https://doi.org/10.1021/acs.analchem.5c03783
文章改编转载自微信公众号:DrugAI
原文链接:https://mp.weixin.qq.com/s/6r82KiENL5w9DO65-SiRvw | 






















