西湖大学原发杰实验室研发的ProTrek蛋白质语言大模型,革新了蛋白质寻找方式。研究者输入自然语言即可精准定位目标蛋白质,将数月筛选缩短至几分钟,还能发现“形不似神似”的优质蛋白质。团队同步推出SaprotHub开源平台,让无编程基础的生物学家也能轻松训练专属模型。两项成果发表于《自然·生物技术》,由博士生粟锦作为第一作者主导,为新药研发与生物技术创新提供强力支撑,开启蛋白质科学“大航海时代”。
蛋白质是生命的基石,寻找具有特定功能的蛋白质,是研发新药、开发新生物技术的起点。然而,在由数百亿蛋白质构成的“宇宙”中找到目标蛋白质,一直是件棘手的事情。
这是因为,以往生物学家寻找蛋白质使用的是传统的实验筛选的方法——就像“以图搜图”,只能根据已知蛋白质的“画像”,也就是其序列或结构去比对相似的新蛋白质。
这就如同大海捞针,不仅效率低下,且可能遗漏掉那些“形不似而神似”的蛋白质,导致许多功能优异的“暗物质”难以被发现。
近日,西湖大学工学院原发杰实验室研发的ProTrek蛋白质语言大模型,让寻找蛋白质这一过程变成了像“百度一下”这么简单。
ProTrek就像一个搜索引擎,研究者只需输入一段描述目标蛋白质特征的自然语言(例如:能够高效切割DNA某一位点的工具),AI就能在浩瀚的蛋白质宇宙中精准定位所需寻找的蛋白质。
以往耗时数月的筛选过程,现在只需几分钟。
不止于“授人以鱼”,团队更进一步“授人以渔”。他们同步推出SaprotHub开源平台,让不具备编程基础的生物学家,根据简单清晰的网页指示,点击几下鼠标即可轻松训练自己所需要的蛋白质语言大模型。
近日,这两项成果本月分别以“A tri-modal protein language model enables advanced protein searches”和“Democratizing protein language model training, sharing and collaboration”为题发表于Nature Biotechnology。

论文链接:https://www.nature.com/articles/s41587-025-02836-0

论文链接:https://www.nature.com/articles/s41587-025-02859-7
寻找蛋白质,就像使用搜索引擎一样简单
两项重磅成果的接连发布,背后是近三年的扎实积累。而这段科研旅程的核心人物之一,是两项成果的第一作者——原发杰实验室的博士生粟锦。
2021年秋天,对粟锦和原发杰教授而言都是个转折点。原发杰刚刚离开腾讯加入西湖大学,亟需组建团队;而粟锦则在寻找能将计算机与生命科学深度融合的研究方向。两人的相遇恰逢其时——粟锦成为了原发杰在西湖大学的第一个博士生,一段“新手导师与新手博士”的科研探索就此开启。
“这正是我一直在寻找的方向。”粟锦回忆道。在深入调研后,他发现蛋白质研究领域存在一个巨大的瓶颈:虽然蛋白质数据库规模爆炸式增长,但科学家们缺乏有效的工具来挖掘其中的宝藏。这个发现让他坚定了研究方向——开发全新的AI方法来破解蛋白质功能预测的难题。
正是基于这样的思考,粟锦在原老师的指导下,开始了ProTrek和SaprotHub的研发。他们的目标很明确:要打造一个能真正“理解”蛋白质的智能系统。

粟锦(左)与论文合作者、实验室同学周禧彬(右)
那么,ProTrek究竟是如何做到的呢?这款“蛋白质宇宙的导航引擎”拥有三大绝活:
首先是“三合一”的理解能力。ProTrek是全球首个能同时理解蛋白质三种核心信息(氨基酸序列、三维空间结构、自然语言功能描述)的模型,它将这三种不同维度的信息融合在一个统一的智能框架里,建立了深度的关联。
其次是“意会”而非“形似”的搜索。正因为有了深度的理解,ProTrek能够进行“功能搜索”。它不再拘泥于序列或结构的局部相似,而是从全局把握蛋白质的核心功能。
最后是百倍速的超级引擎。研究团队基于ProTrek搭建的线上服务器,收录了超过50亿个蛋白质的数据,检索速度比传统工具快上百倍,几分钟内就能完成对整个数据库的扫描。
在工科实验室诞生的ProTrek,发现蛋白质的能力究竟如何?去生科实验室“遛一遛”就知道了。
生命科学学院常兴实验室的博士后何燕,正在测试一种由ProTrek从包含2亿个蛋白质的数据库中搜索到的,与人类尿嘧啶DNA糖基化酶(UDG)功能相似的新蛋白质V1。

何燕
实验结果令人兴奋:新发现的V1蛋白经基因编辑实验验证,比现有蛋白质的基因编辑效率更胜一筹,操作精准度也显著提升。
训练大模型,就像使用App一样轻松
如果说ProTrek是为无垠蛋白质宇宙探索者准备的“星际导航系统”,那么SaprotHub则像一个工作坊,探索者可以在其中根据自己的需求,创造专属的探索工具。
为什么想到研发SaprotHub这样一个开源平台?粟锦深知训练大模型的过程有多复杂。即便是对于他这样计算机科班出身的人来说,从编程环境配置、海量数据预处理到模型训练和评估,每个步骤都需付出大量的努力。而对于没有计算机基础的生物学家来说,这项任务更是难上加难。

粟锦
然而,AI技术发展的意义,并非设置更多“黑科技”的门槛,而是让科技变得更加民主和普及,令更多人能享有技术带来的便利。
于是,几乎在研发ProTrek的同时,原发杰团队启动了SaprotHub的研发。
SaprotHub的核心引擎是Saprot语言模型,它开创性地提出了一种“结构感知”(Structure-Aware)词汇表,将蛋白质的一维氨基酸序列与其三维局部结构信息进行联合编码,构建出一种全新的蛋白质“语言”。
为了让生物学家也能够拥有训练前沿蛋白质语言模型的能力,团队基于免费的Google Colab云平台,通过数月开发,上万行代码的编写,开发了ColabSaprot“一键式”开源训练平台。
在这一平台上,生物学家只需按照网页提示简单操作,即可轻松地训练蛋白质语言模型,实现从想法到验证的快速迭代。举个例子,一位研究癌症靶点的医学专家,在半小时内,不需要学习任何编程,就能根据自己的实验数据训练出定制化的AI模型。
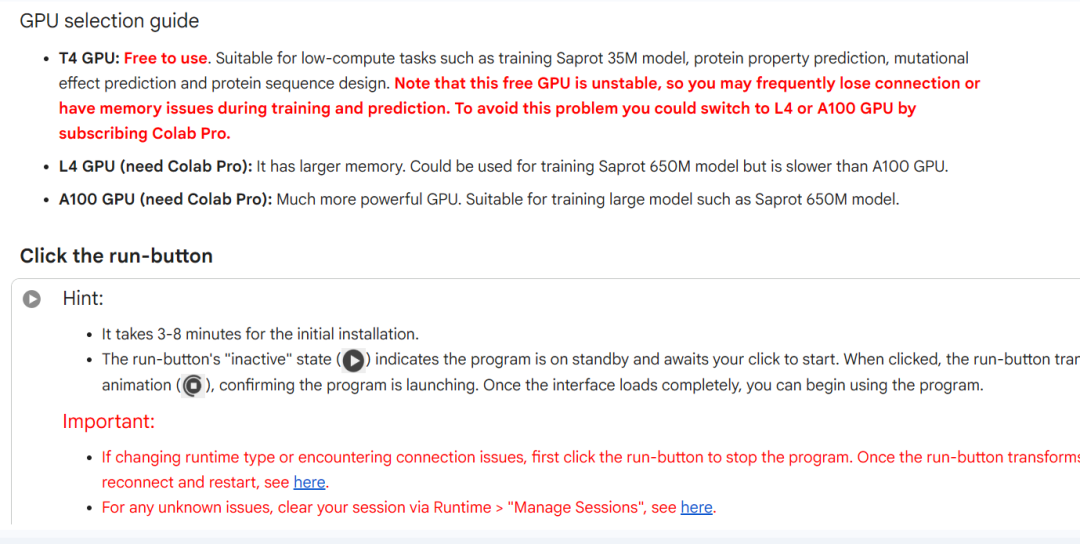
ColabSaprot开源训练平台界面
从写繁琐的代码,到只需轻点几次鼠标可操作,SaprotHub将复杂的“授人以渔”过程转化为一份简单的“使用说明书”。团队录制了详细的使用教程,从模型训练到使用,手把手教学,真正实现了“开箱即用”。
这个反复打磨的过程,正是科技走向普及的必由之路——将复杂留给自己,将简单留给使用者。
SaprotHub平台不仅操作简便,其预测有效性也在一系列计算机模拟性能实验和生物湿实验中得到了验证。在原发杰团队开展的用户研究中,12位没有AI背景的生物学研究者使用该平台时,取得了与AI研究者相媲美的成果;一家生物技术公司利用该平台对工业用木聚糖酶进行改造,成功将酶的活性提升了2.55倍;研究人员对基因编辑工具进行优化,预测出的新版本在实验中展现出翻倍的编辑效率;平台还被用于设计更亮的绿色荧光蛋白(GFP),其中一个新设计的蛋白的荧光亮度达到了原始版本的8倍以上 。
SaprotHub还设有开放蛋白质模型联盟(OPMC)成员共建社区,汇聚了来自MIT、哈佛、牛津等全球顶尖机构的研究力量。研究者们可以像在应用商店里一样,分享和使用彼此的模型,形成了一个良性循环的创新生态。
ProTrek入口(硬件升级中,预计11月1日开放)
search-protrek.com
SaprotHub入口,请点击文末“阅读原文”。
从此,不具备编程背景的生物学家,也能从AI的“使用者”转变为“创造者”和“贡献者”。
原发杰实验室的两项突破性进展,赋予了蛋白质宇宙以星图和罗盘,更将探索的罗盘交到每一位研究者手中——一个真正意义上的蛋白质科学的“大航海时代”已经到来。
致谢
A tri-modal protein language model enables advanced protein searches
本研究由西湖大学原发杰实验室、常兴实验室与香港科技大学卢泓远教授团队主导完成,粟锦、何燕和游世洋为共同第一作者。项目得到2Beltrao、Ovchinnikov、Zeng、Wang、Huang等多位国内外学者的建议与指导。感谢L. Hong与M. Li提供基础数据库支持,感谢西湖大学高性能计算中心(HPC Center)在算力与技术上的持续保障。本研究受浙江省“领雁计划”、国家自然科学基金、国家重点研发计划、国家科技重大专项、广州市产学研联合资金项目以及西湖大学合成生物与集成生物工程研究中心等的资助与支持。本研究感谢所有参与者在模型算法开发、实验验证及论文修改中的共同努力。
Democratizing protein language model training, sharing and collaboration
本研究由西湖大学原发杰实验室联合多家高校与研究机构开展完成。粟锦为论文第一作者,李志凯、陶天立、韩晨晨、何燕等为主要贡献者,Steinegger、Ovchinnikov等国际学者在模型设计和论文修订中提供了重要指导。 研究工作获得国家重点研发计划、国家自然科学基金、韩国国家研究基金、Novo Nordisk基金会、浙江省“领雁计划”、西湖大学未来产业研究中心及浙江省低碳智能合成生物学重点实验室等项目支持。感谢西湖大学HPC中心及来自首尔大学、哈佛大学、麻省理工学院等合作团队在模型训练、实验验证、共建平台和论文撰写中的协同支持。
本文转自【西湖大学WestlakeUniversity】公众号
文章改编转载自微信公众号:智药邦
原文链接:https://mp.weixin.qq.com/s/Z6Mlr1Uayd--dk8s9iPiFQ | 











