2025 年发表于《Nature Communications》的研究,提出面向基因组序列理解的生成式基础模型 OmniReg-GPT。该模型通过优化注意力机制,突破传统 Transformer 瓶颈,可高效处理 50k–100k bp 长序列,显著降低训练显存与速度需求。预训练阶段能捕获单核苷酸至百万碱基尺度的调控元件分布,在顺式调控元件识别、eQTL 效应预测、单细胞染色质可及性分析等多下游任务中表现卓越,且可通过提示工程生成细胞类型特异性候选增强子。OmniReg-GPT 为多尺度基因调控研究提供了统一高效的新范式,拓展了基因组基础模型的能力边界。
人类基因组包含大量控制基因活性与生物功能的复杂调控元件。构建能够高效处理长序列输入的大窗口基础模型,是理解多层级、复杂顺式调控景观的关键挑战。研究人员提出 OmniReg-GPT,这是一种使用优化注意力机制、可在低资源条件下完成长基因组序列预训练的生成式基础模型。在预训练过程中,OmniReg-GPT 能以高效率捕获从单核苷酸到百万碱基尺度的调控元件分布,并显著降低训练速度与显存需求。
在多种下游任务中,研究人员证明 OmniReg-GPT 在不同尺度的调控应用中均展现卓越性能,包括多类型顺式调控元件识别、上下文依赖的基因表达预测、单细胞染色质可及性分析以及三维染色质接触建模。此外,作为生成模型,它还能通过提示工程生成细胞类型特异性的候选增强子。OmniReg-GPT 扩展了基因组基础模型的能力边界,并为基因组研究提供了重要的预训练资源。

长期以来,基因组学的核心目标是理解基因组序列的“语言”,尤其是数量占据绝大部分的非编码调控序列。虽然蛋白编码基因只占基因组极小部分,大量非编码区域却以高度复杂的方式参与基因调控,例如通过时空与组合方式共同调节 RNA 聚合酶的招募和活性。
随着多种功能基因组测序技术的发展,大型国际计划已经系统性绘制了不同细胞环境下的调控基因组,并识别出数量庞大的调控元件(如增强子、启动子、沉默子、绝缘子等)。基于这些数据,监督式深度学习模型在基因组领域崭露头角,可实现 DNA 序列和功能数据之间的映射,推动了顺式调控注释、基因表达建模和变异效应预测等应用。
然而,这些模型往往依赖于特定功能数据用于训练,导致出现大量为不同预测目标设计的专用模型,难以形成全面、统一的调控序列表示。同时,由于 Transformer 自注意力机制的计算与显存开销随序列长度平方增长,大多数 DNA 预训练模型只能处理短序列,导致对真实调控序列(跨越成千上万乃至百万碱基)的理解十分有限。
这些挑战凸显出:必须开发具备更大窗口、更高效率、能够建模长程依赖的序列基础模型。
方法
研究人员设计了 OmniReg-GPT,这是一种可处理极长基因组序列的生成式基础模型。其核心是经过优化的注意力机制,可以大幅降低处理长序列的计算与内存成本,使模型能够在低资源条件下完成预训练。模型在长序列片段上通过自监督学习方式,构建从核苷酸到百万碱基范围的多尺度表示。随后,研究人员在广泛的下游任务中利用统一框架对模型进行微调或评估其零样本能力,以全面验证 OmniReg-GPT 在调控序列理解上的潜力。
结果
OmniReg-GPT 的模型架构与效率优势
OmniReg-GPT 采用高效注意力机制,可处理高达 50k–100k bp 的长序列输入。
· 模型的显存使用量随序列长度的增长明显低于最新 Transformer 模型,可在一般 GPU(如 32GB V100)上顺利运行。
· 计算速度也显著优于传统长序列 Transformer 模型,在 10k–50k bp 区间均保持较低的时间消耗。
这些结果表明,优化后的注意力机制使 OmniReg-GPT 在长序列处理方面具备极强的可扩展性。

预训练阶段学习多尺度调控分布
研究人员在分析预训练表征后发现:
· 模型能够在无监督条件下学习不同尺度的调控规律,包括启动子、增强子、绝缘子等信号的分布规律。
· OmniReg-GPT 对较短序列的局部调控模式和跨越更长距离的调控互动均展现出良好识别能力。
这种跨尺度感知能力,使模型能够同时捕捉局部序列语法与远程调控关系。
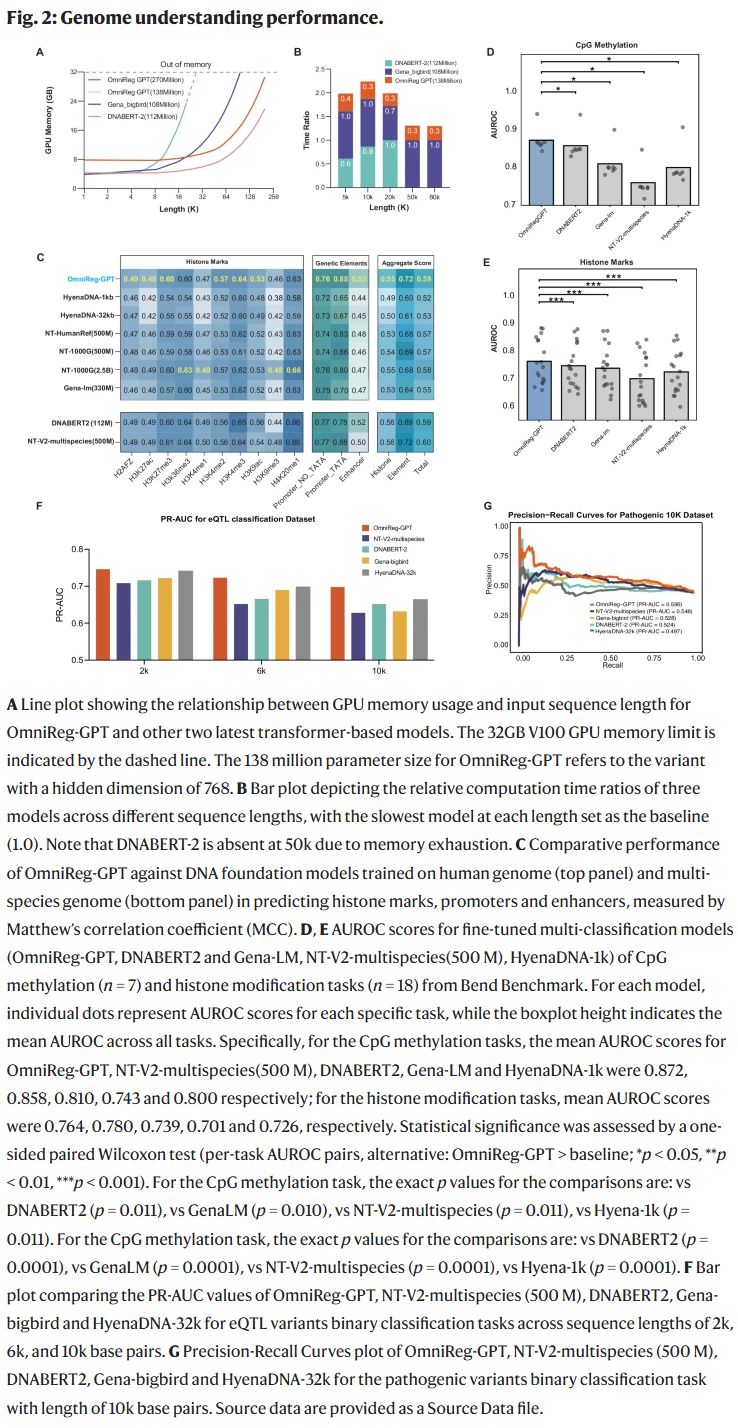
在多类调控元件预测中表现优异
研究人员对比了 OmniReg-GPT 与其他 DNA 基础模型在多项调控任务上的表现(如组蛋白修饰、启动子与增强子预测):
· OmniReg-GPT 在大多数任务中的 MCC 指标均达到最高或接近最高水平。
· 特别是在特定转录因子结合预测任务中,模型的表现明显优于 DNABERT-2。
这表明模型对调控序列的功能特征建模更为充分。
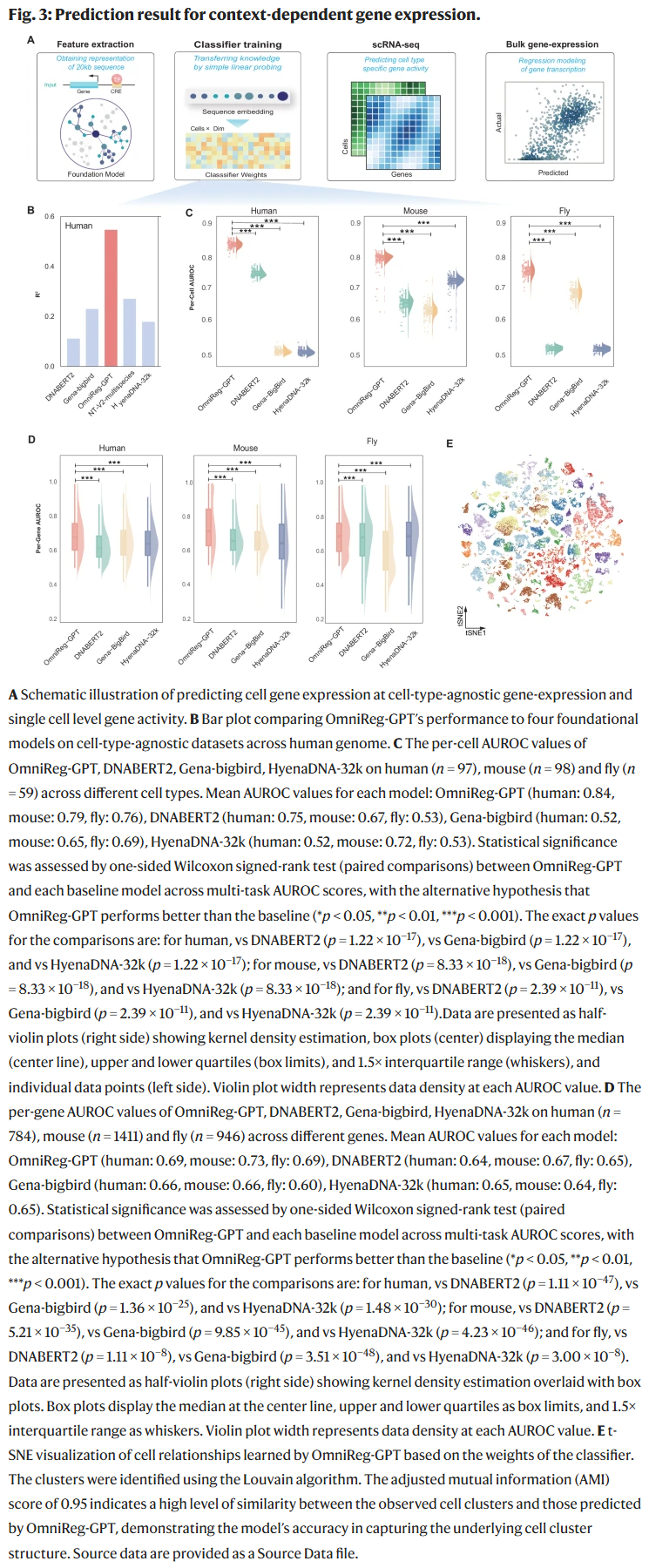
更准确地预测 eQTL 与致病变异效应
模型在两个重要任务中均显著领先:
eQTL 效应预测
在 2k、6k、10k bp 长度下,OmniReg-GPT 的 AUROC 均为最高(0.724 / 0.719 / 0.701)【turn2file2†L21-L24】。
致病 SNP 区分
OmniReg-GPT 在 10k 序列上获得 AUROC = 0.679,远高于其他模型(如 NT-V2、DNABERT-2 等)【turn2file2†L25-L27】。
结果显示模型对变异功能效应具有良好的敏感性和泛化能力。

上下文依赖的基因表达预测性能显著提升
基因表达由复杂的上下文语法控制,需要模型理解更大范围的调控背景。研究人员在更广序列窗口的背景下评估基因表达预测能力:
· OmniReg-GPT 能更准确建模远程调控元件对目标基因的综合影响。
· 在不同细胞类型和基因组范围的测试中,模型表现均优于其他基础模型【turn2file2†L36-L42】。
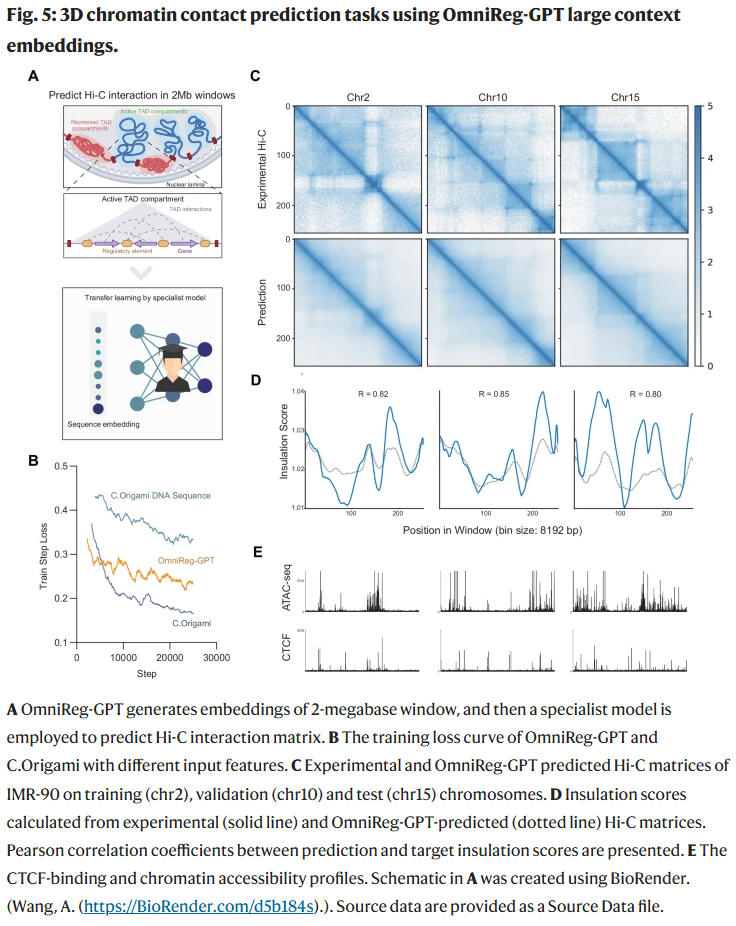
单细胞染色质可及性与三维染色质结构建模
单细胞 ATAC-seq 模型
研究人员固定模型主干,仅训练简单线性层评估模型的表示质量【turn2file4†L3-L9, L10-L23】:
· OmniReg-GPT 生成的细胞嵌入在 kNN 聚类中得到与真实细胞类型高度一致的分群结构。
· 在 ARI、AMI 与同质性指标上均超过 snapATAC、chromVAR 等工具。
三维染色质接触预测
研究人员进一步使用模型输出预测跨基因组尺度的染色质接触强度,实现对 3D 基因组折叠模式的建模。结果显示模型具备对跨巨量序列的立体调控关系进行推断的能力。
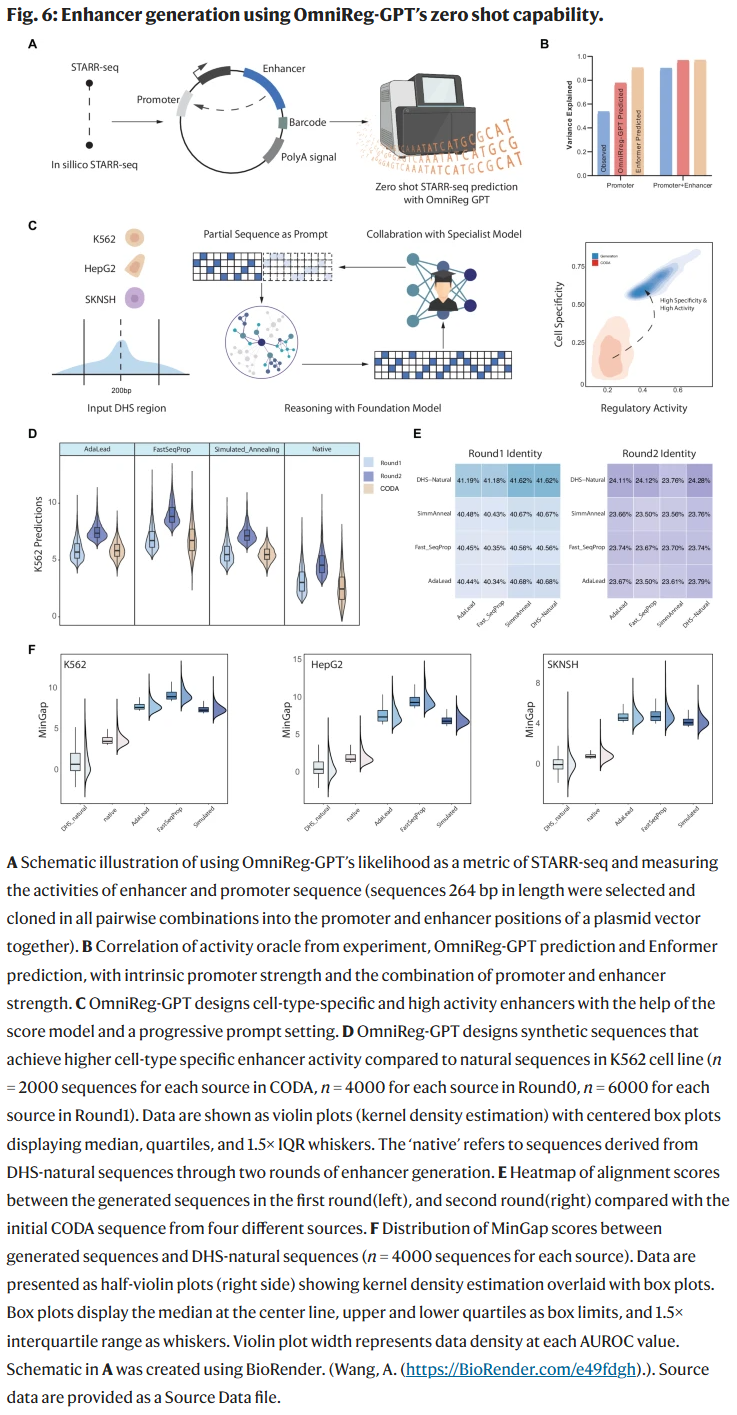
讨论
OmniReg-GPT 展示了大窗口高效基础模型在基因组学中的巨大潜力。其主要贡献包括:
突破长序列建模瓶颈
通过优化注意力机制,模型可在普通显存环境中处理极长序列,为大尺度调控建模奠定基础。
实现跨尺度序列理解
从单核苷酸到百万碱基的调控规律均可有效捕获,避免过去模型依赖短序列造成的信息缺失。
在广泛下游任务中表现强劲
包括调控元件识别、变异效应预测、基因表达建模、单细胞分析及 3D 基因组结构推断。
生成式能力开拓新方向
通过提示工程,模型可生成细胞类型特异的候选增强子,为实验设计与功能验证提供新工具。
总体来看,研究人员的工作展示了基础模型在基因组调控层面实现“全面理解”的可行性,并为多尺度基因调控研究提供了重要的新范式。
参考资料
Wang, A., Li, J., Dong, H. et al. Omnireg-gpt: a high-efficiency foundation model for comprehensive genomic sequence understanding. Nat Commun 16, 10139 (2025).
https://doi.org/10.1038/s41467-025-65066-7
文章改编转载自微信公众号:DrugOne
原文链接:https://mp.weixin.qq.com/s/DjsC8z2Fke7iDiOLQ7j1Uw?scene=1 | 











