本帖最后由 graphite 于 2025-12-27 02:01 编辑
多国联合团队2025年在 Nature Communications 发表的《 Designing molecular RNA switches with Restricted Boltzmann machines 》提出基于受限玻尔兹曼机(RBM)的 RNA 开关设计方法。RBM 从天然 SAM-I 核糖开关序列中学习规律,生成 476 条人工序列,部分与天然序列差异达 40%。经 SHAPE 和 DMS 实验验证,RBM 高评分(>300)序列对 SAM 的结构开关响应成功率达 35%,且能建模假结等关键三级结构。该方法还适配环二腺苷酸等其他核糖开关家族,为合成生物学调控工具开发提供新路径。

在细菌的基因调控中,有一种神奇的 “分子开关”—— 核糖开关(Riboswitch)。它无需蛋白质协助,仅靠自身 RNA 结构变化,就能感知代谢物浓度并调控基因表达,是生命体内高效的 “天然传感器”。然而,人工设计这种能精准响应特定分子的 RNA 开关,一直是合成生物学的难题 —— 传统方法难以同时兼顾结构稳定性与开关灵活性。2025 年,法国、德国联合团队在《Nature Communications》发表重磅研究,利用受限玻尔兹曼机(RBM) 构建生成模型,成功设计出功能与天然核糖开关相当的人工 RNA 分子,部分序列与天然序列差异达 40% 仍能高效响应,为基因编辑、代谢工程等领域提供了全新工具。
一、天然 RNA 开关的 “精妙之处” 与人工设计的 “拦路虎”
要理解人工设计的难点,首先得看清天然核糖开关的 “工作原理”:
1.1 天然核糖开关的双重身份

图1 SAM-I 核糖开关的结构二态性(左:ON 状态,右:OFF 状态,结合 SAM 后形成假结与终止子结构)
核糖开关的核心是适配体结构域(Aptamer),它能实现 “结构二态性”:
无代谢物时(ON 状态):适配体结构松散,下游基因的转录或翻译不受阻,基因正常表达;
结合代谢物后(OFF 状态):适配体与代谢物(如 S - 腺苷甲硫氨酸 SAM)结合,触发构象折叠,形成终止子结构,阻断基因表达(如图 1)。
这种 “感知 - 响应” 的全 RNA 自主机制,使其成为合成生物学中理想的调控工具,但天然核糖开关的序列与结构高度耦合,人工复刻难度极大。
1.2 人工设计的三大挑战
长期以来,人工设计 RNA 开关主要面临三大障碍:
结构 - 功能映射复杂:RNA 序列与三维结构的关系是非线性的,微小的序列变化可能导致功能完全丧失,传统热力学模型难以精准预测;
三级结构难兼顾:核糖开关的功能依赖假结(Pseudoknot)等三级结构,而传统设计方法(如协方差模型 CM)只能捕捉二级结构,无法建模这些关键相互作用;
开关灵活性不足:人工设计的 RNA 常出现 “锁死” 在单一构象的问题,无法像天然分子那样在 “开放 - 闭合” 状态间灵活切换。
以 SAM-I 型核糖开关为例,它需要结合 SAM 后稳定 P1 螺旋和假结结构,这种协同折叠的复杂性,让人工设计的成功率长期低于 10%。
二、RBM 的 “魔法”:从天然序列中学会设计密码
研究团队的核心创新,是用 RBM 从天然核糖开关序列中 “学习” 设计规律,而非直接模拟生物物理过程。RBM 是一种两层生成式神经网络,能捕捉数据中的统计规律,尤其擅长建模序列间的关联与变异:
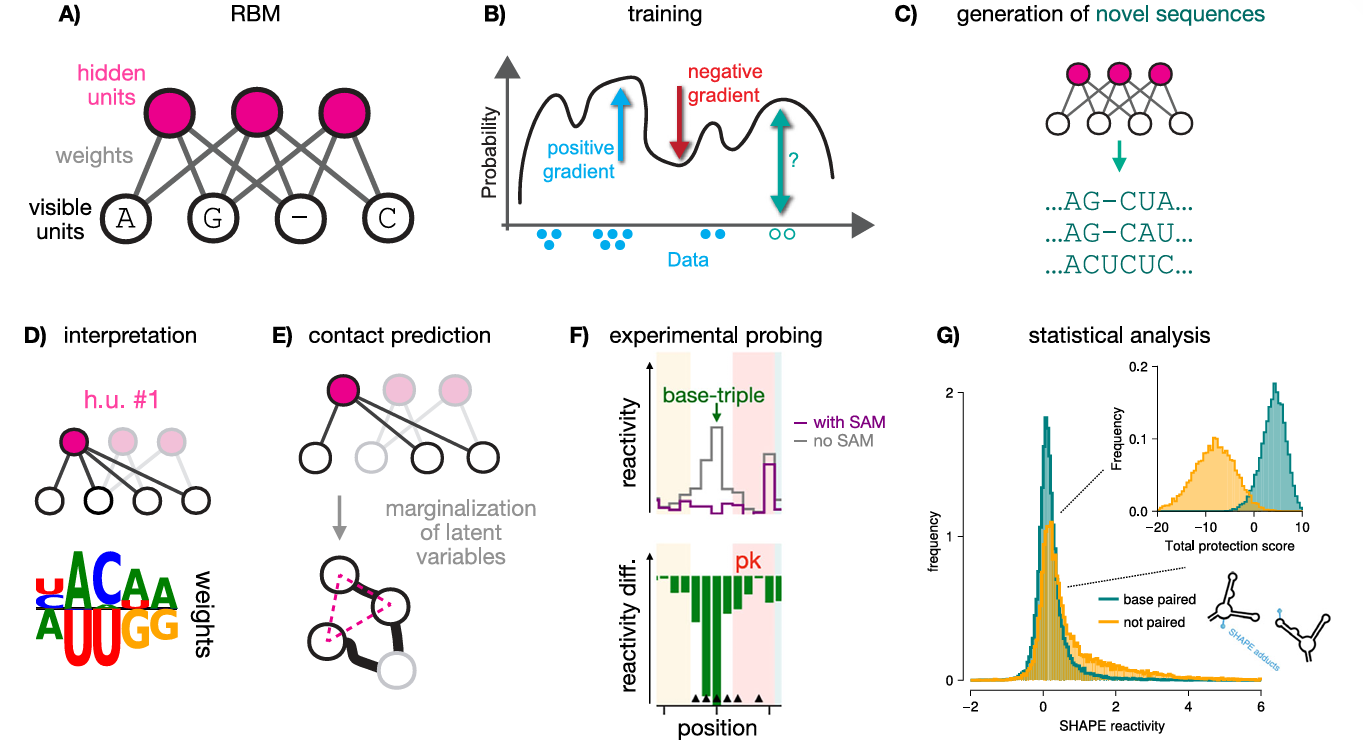
图2 基于 RBM 的 RNA 开关设计与验证流程
2.1 训练过程:让 AI “读懂” 天然 RNA 的进化密码
团队首先收集了 Rfam 数据库中 SAM-I 型核糖开关的同源序列,构建多序列比对(MSA)数据集。将这些序列输入 RBM 后,模型通过两个关键步骤学习:
特征提取:可见层(Visible Layer)编码 RNA 序列的核苷酸(A/C/G/U/ 间隙),隐藏层(Hidden Layer)自动提取潜在特征,比如 “哪些位置的核苷酸必须互补以形成螺旋”“假结结构需要哪些序列模式支撑”;
规律建模:通过最大化正则似然函数,RBM 学习天然序列的保守性(如关键结合位点的核苷酸不变性)与协变性(如螺旋区的碱基互补配对变异),最终形成一个能生成 “符合功能规律” 的 RNA 序列的概率模型。
与传统协方差模型(CM)相比,RBM 的优势在于能捕捉高阶相互作用—— 不仅能建模碱基对之间的二级结构关联,还能识别假结中三个及以上核苷酸的协同关系,这正是天然核糖开关功能的核心。
2.2 RBM 的核心公式:量化序列与结构的关联
RBM 的生成能力源于其对序列概率分布的精准建模,核心公式贯穿 “能量计算 - 概率转化 - 特征整合” 全流程:
联合能量函数:定义可见层(RNA 序列 v)与隐藏层(潜在特征 h)的联合能量,量化两者的相互作用:
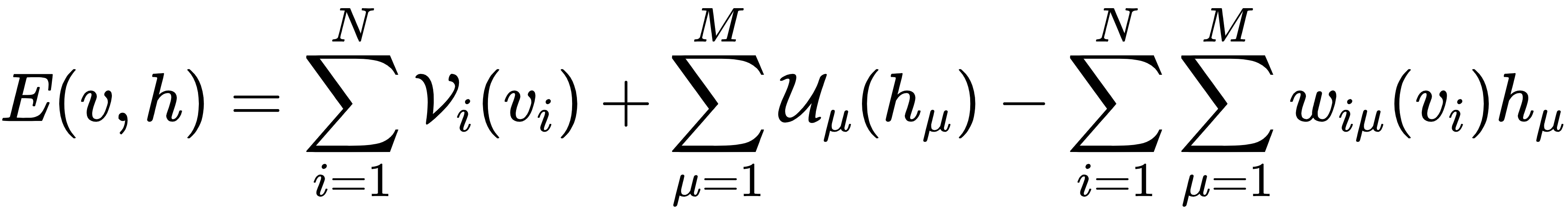
其中,Vi(vi)是可见层核苷酸的偏置势能,Uμ(hμ)是隐藏层单元的偏置势能,wiμ(vi)是两层间的交互权重,N 为序列长度,M 为隐藏单元数。
序列概率分布:通过玻尔兹曼分布,将能量转化为序列出现的概率,能量越低的序列越可能具备功能:

Z 为归一化配分函数,Eeff(v)是边际化隐藏变量后的有效能量,可高效计算无需遍历所有隐藏状态,这也是 RBM 能快速生成候选序列的关键。
保护分数(Protection Score):后续实验验证中,用于量化 RNA 位点的配对概率,判断结构开关是否生效:
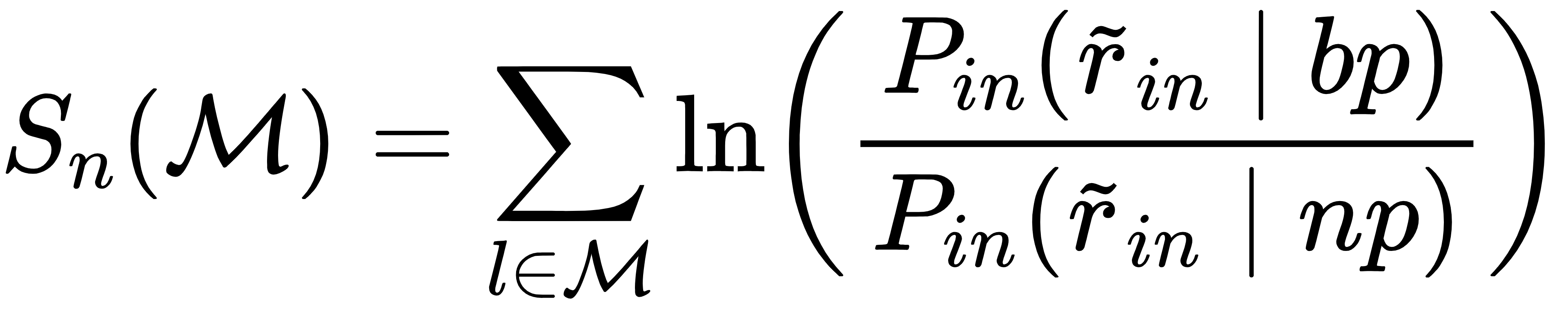
其中M是关键位点集合(如假结、SAM 结合位点),P(~rin|bp)和P(~rin|np)分别是位点配对与未配对时的反应活性概率,分数正负变化直接反映构象切换。
2.3 设计流程:从模型采样到功能 RNA
训练完成后,团队通过两个筛选步骤从 RBM 生成的序列中获得候选分子:
分数筛选:保留 RBM 评分高于 300 的序列(该分数反映序列与天然家族的契合度);
多样性筛选:优先选择与天然序列差异较大(10%-40%)的序列,验证模型的泛化能力。
最终,团队设计了 476 条人工 RNA 序列,与 201 条天然序列一同进行实验验证。
三、实验验证:人工 RNA 开关的 “实战表现”
为了检验人工 RNA 的功能,团队采用两种化学探测技术(SHAPE-MaP 和 DMS),通过检测 RNA 核苷酸的反应活性变化,判断其结合代谢物后的构象转换:
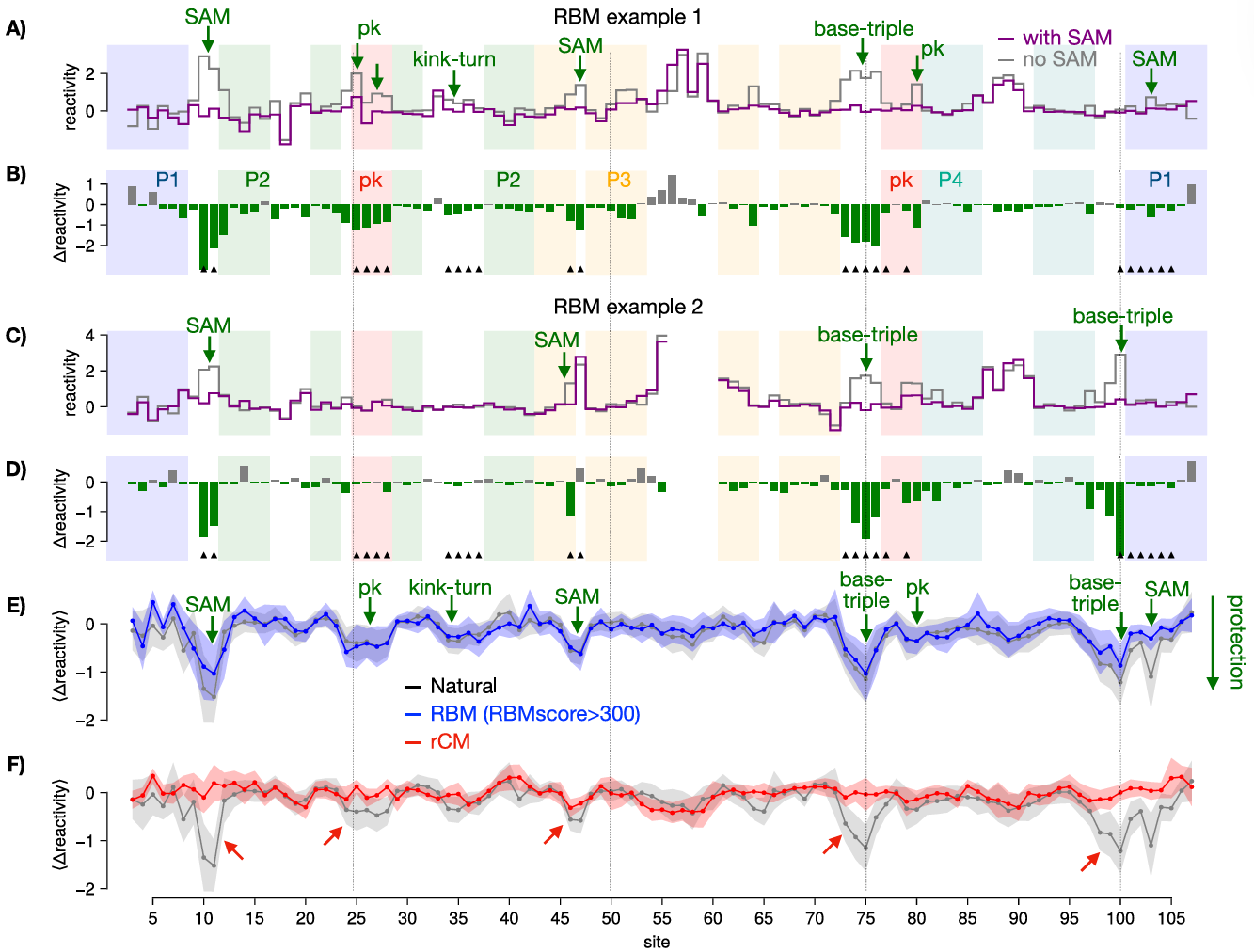
图3 RBM 设计 RNA 与天然 RNA 的 SHAPE 反应活性对比
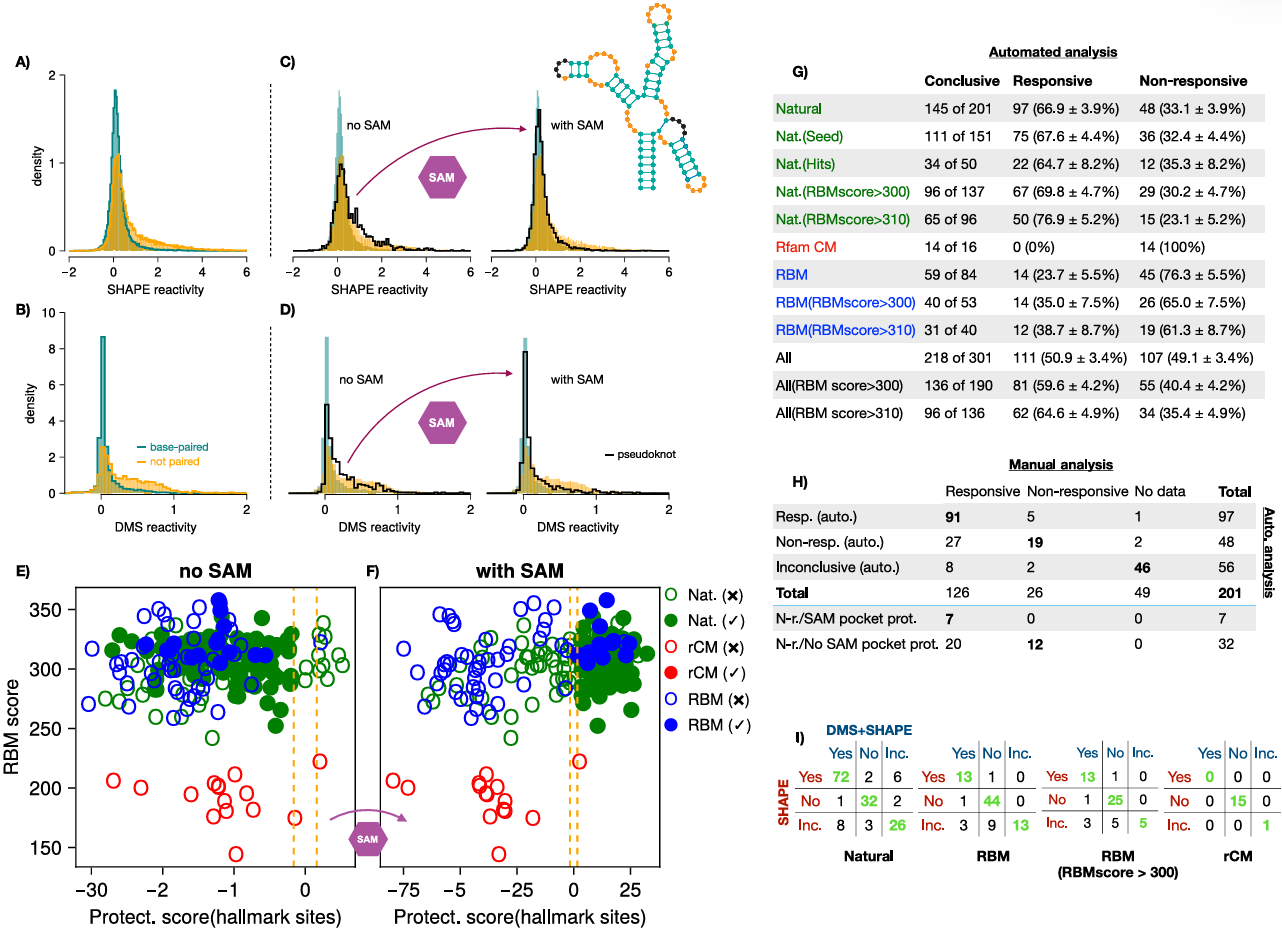
图4 天然 RNA 与不同模型设计 RNA 的功能成功率对比
3.1 结构开关验证:与天然分子 “行为一致”
化学探测的核心逻辑是:RNA 中未配对的核苷酸反应活性高,配对或结合代谢物后活性降低(即 “保护效应”)。实验发现:
高评分的人工 RNA 序列(RBM 分数 > 300)与天然序列表现出高度相似的保护模式 —— 结合 SAM 后,假结区(25-28 位、77-80 位)、SAM 结合位点(10 位、11 位等)的反应活性显著下降,证明其成功折叠为闭合构象;
部分人工序列即使缺失天然序列中的 P4 螺旋结构,仍能正常响应 SAM,展现出比天然分子更强的结构灵活性。
3.2 功能效率:35% 的成功率媲美天然开关
通过统计 “保护分数”(衡量关键位点构象变化的显著性),团队得出核心结论:
在结论明确的人工序列中,35% 能高效响应 SAM,与天然核糖开关的功能成功率(66.9%)虽有差距,但考虑到人工序列与天然序列的差异,这一结果已远超传统设计方法;
当人工序列与天然序列差异在 20% 以内时,功能成功率高达 50%,部分序列甚至能在 SAM 浓度仅 0.1mM 时触发开关;
对比实验显示,传统协方差模型(CM)设计的序列无一提振功能,证明 RBM 对三级结构的建模能力是成功的关键。
3.3 关键发现:开关功能的 “能量窗口”

图5 RNA 开关功能的能量窗口
团队还发现一个重要规律:人工 RNA 开关的功能与 P1 螺旋、假结的配对能量密切相关 —— 只有当这些结构的形成能量处于 -10~0 kcal/mol 的中间区间时,才能实现灵活开关。若能量过低(结构过稳定),RNA 会 “锁死” 在闭合状态;若能量过高(结构过松散),则无法稳定结合代谢物。而 RBM 生成的序列,恰好优先落在这个 “功能能量窗口” 内,这正是其设计成功率高的核心原因。
四、RBM 设计的 “独特优势”:超越传统方法的核心逻辑
与传统 RNA 设计方法相比,RBM 的生成式建模思路具有三大不可替代的优势:
4.1 从 “规则驱动” 到 “数据驱动”
传统方法依赖已知的 RNA 结构规则(如碱基配对原则),而 RBM 直接从天然序列数据中学习规律,能捕捉人类尚未发现的潜在模式 —— 例如某些看似不保守的位点,其实是维持开关灵活性的关键,这种 “数据自主发现” 让设计更贴近自然规律。
4.2 可解释性与创造性兼备
RBM 的隐藏层权重可可视化,能直接揭示功能相关的序列基序(Motif):例如某一隐藏单元的权重分布显示,它同时调控 P1 螺旋的碱基互补和假结的形成,证明这两个结构的协同稳定对开关功能至关重要(如图 3)。这种可解释性,让研究人员能反向优化设计策略。
4.3 泛化能力强,适配多样需求
RBM 不仅能设计 SAM 响应型开关,团队还验证了其在环二腺苷酸(c-di-AMP)、甘氨酸等其他核糖开关家族的适用性。此外,通过调整模型参数,还能设计出缺失特定螺旋(如 P4)的极简开关,满足不同场景的应用需求。
五、应用前景:从实验室工具到产业级应用
人工 RNA 开关的成功设计,为多个领域打开了想象空间:
5.1 精准基因调控工具
将人工 RNA 开关与编辑工具(如 CRISPR)结合,可实现 “代谢物触发的基因编辑”—— 例如在肿瘤细胞中,当检测到异常高浓度的代谢物(如乳酸)时,才启动编辑程序,降低脱靶风险。
5.2 代谢工程优化
在微生物发酵中,可设计响应目标产物(如乙醇、氨基酸)的 RNA 开关,当产物浓度达到阈值时自动关闭合成通路,避免产物过量抑制生长,提升发酵效率。
5.3 生命起源研究
RNA 开关被认为是 “RNA 世界假说” 的关键证据,人工设计的功能 RNA 开关,为研究早期生命如何通过 RNA 实现自主调控提供了绝佳的实验模型。
六、总结
这项研究的核心价值,在于首次用生成式 AI 突破了 RNA 开关设计的 “结构 - 功能” 瓶颈。RBM 不再局限于模拟已知的生物物理规则,而是通过学习天然序列的进化信息,自主探索功能 RNA 的序列空间,甚至设计出天然中不存在但功能更优的分子。随着技术迭代,未来可能实现 “定制化 RNA 开关”—— 输入目标代谢物和调控需求,AI 就能快速生成对应的 RNA 序列,为合成生物学、基因治疗、生物制造等领域带来革命性变化
原文链接:https://www.nature.com/articles/s41467-025-66265-y | 


























