本帖最后由 哈奇一 于 2026-1-27 23:12 编辑
2026年1月9日,弗吉尼亚大学研究人员在《Journal of Chemical Information and Modeling》上发表文章,题为“Multimodal Bond Reconstruction toward Generative Molecular Design”。
作者提出了一种多模态图神经网络框架YuelBond,能够在三类关键场景下实现稳健且准确的化学键重建。实验结果表明,YuelBond即使面对不完美或存在噪声的分子数据,依然可以可靠地恢复正确的化学键结构,从而有效弥补了生成式药物发现流程中长期存在的关键缺口。
YuelBond代码仓库:https://github.com/dokhlab/yuel_bond
背景
近年来,生成式人工智能,尤其是生成对抗网络(GANs)和扩散模型,凭借其快速生成海量新颖分子结构的能力,正在深刻改变从头药物发现的研究范式。然而,尽管这些方法展现出巨大潜力,它们仍面临关键性局限,尤其是在化学键级准确性方面。具体来说,在2D分子生成中,预测的键级可能在化学上不合理;而在3D分子生成中,模型通常只输出原子坐标,缺乏明确的化学键注释。因而会带来两个主要问题:(1)分子有效性难以验证,即错误的键级会阻碍对分子有效性的可靠评估;(2)下游任务性能显著下降,包括分子动力学模拟、分子对接以及蛋白–小分子结合预测等。化学键级错误会进一步传递到力场参数化和结合亲和力计算中,导致结果偏差。解决上述问题,是弥合计算机生成分子与真实药物开发应用之间鸿沟的关键。
传统上,化学键级通常通过基于键长和键角的杂化分析,或将原子对与已有化学数据库进行比对来确定,也可以依赖经验性的化学规则或长度规则进行赋值。作为最常用的化学信息学工具包,RDKit通过其xyz2mol程序从三维坐标中推断分子的化学键级。然而,这类基于规则的方法对生成分子中常见的几何畸变并不稳健。当几何结构发生畸变,即键长和键角偏离理想值时,甚至连原子间的连通性判定都会变得困难,而在此条件下预测键级则更加具有挑战性。在生成模型尚未被广泛应用于药物发现之前,这一问题并不突出;但随着生成式模型的快速发展,其影响正变得愈发显著。一方面,生成模型需要不断提高三维结构的几何精度;另一方面,也亟需更加稳健的方法,能够直接从存在畸变的3D坐标中推断分子连通关系和化学键级。
结果
从精确的三维坐标中重建化学键
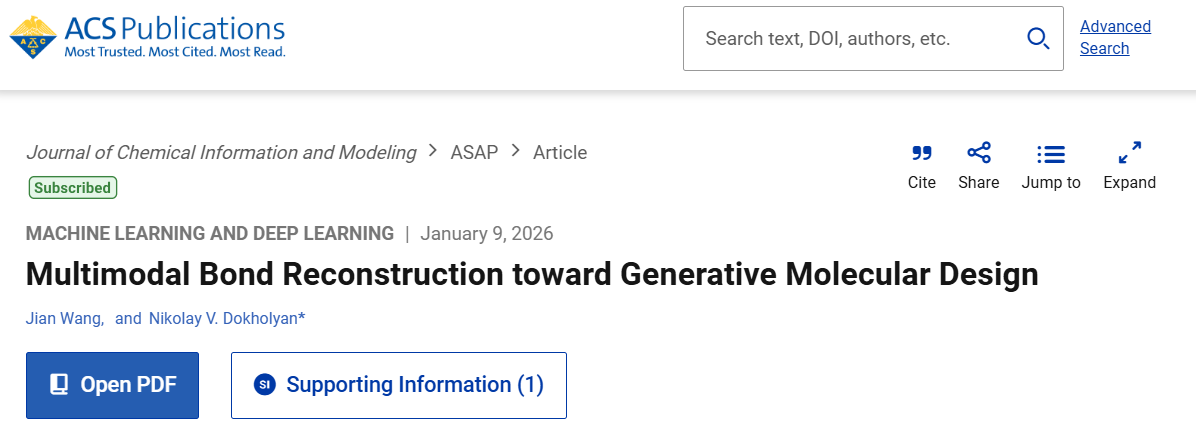
YuelBond基于图神经网络(GNN)构建,对于每个原子,计算其与分子中所有其他原子的距离,并将距离在3Å以内的任意一对原子通过一条边相连(图1),这些成对距离被作为边特征输入模型。作者修改了用于边更新的聚合机制,与仅依赖相连节点特征拼接的方式不同,在更新函数中额外引入了原子间距离以及上一层的边特征。该设计使模型能够更有效地捕捉每一条化学键周围的局部化学环境。随着每一层消息传递的进行,边特征不断被细化,其感受野也随之扩大。在最终阶段,一个线性层将更新后的边特征映射到四种化学键级类别之一:单键、双键、芳香键或三键(图2)。
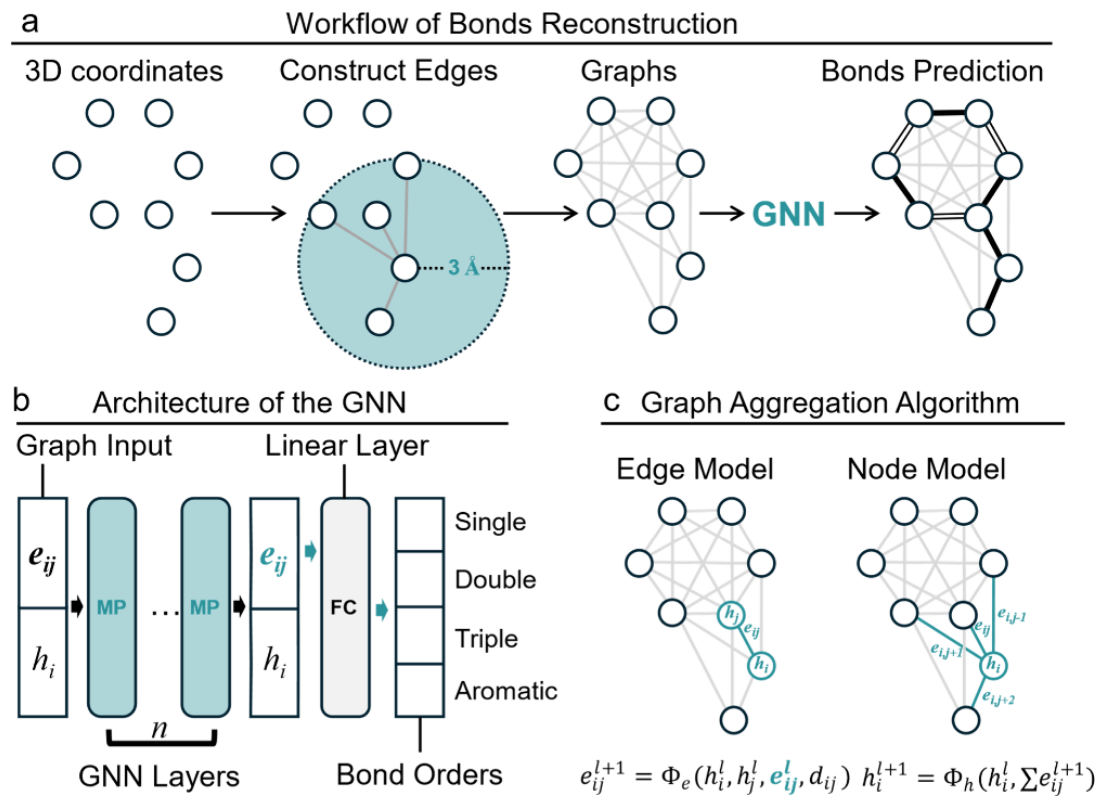
图1 基于GNN的化学键重建流程与架构
使用Geometric Ensemble of Molecules(GEOM)数据集对YuelBond进行了训练和评估,该数据集包含超过45万个分子结构。为评估化学键重建性能,首先移除了分子中的所有键注释,仅基于其三维原子坐标使用YuelBond进行化学键重建,随后在测试集上进行评估。模型取得了98.2%的平均准确率、98.8%的精确率、98.2%的召回率以及98.4%的F1分数,表明其在恢复分子成键信息方面具有很强的能力(图2a)。
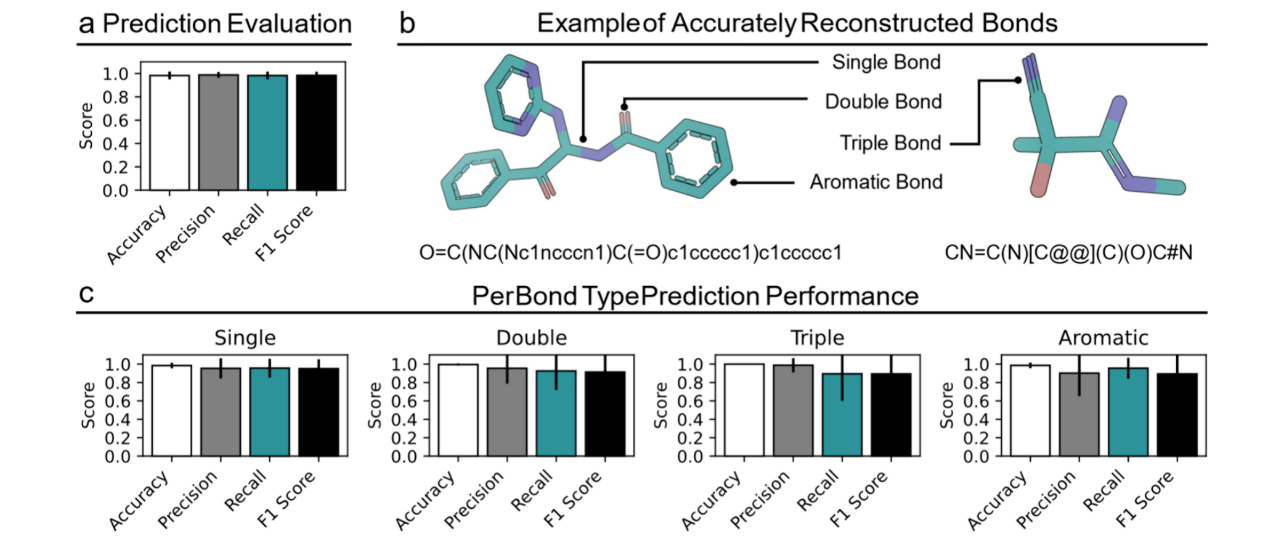
图2 分子结构中化学键级预测性能评估
进一步分析了YuelBond在不同键类型上的预测性能(图2c)。在单键和双键上的高性能表明,该模型能够有效学习常见共价键所对应的典型原子间距离和局部环境。尽管三键预测具有较高的精确率,其召回率略低,这说明模型在赋予三键时表现得较为保守,可能源于三键在数据中的稀有性以及其识别所需的严格几何约束。这种保守性是有益的,因为将非三键误判为三键可能会在下游任务中引入更大的误差。
从粗糙的从头生成化合物中重建化学键
本工作的目标是开发一种能够在粗糙的从头生成化合物(CDG)中重建化学键的模型。这类化合物通常具有噪声较大或不够精确的三维结构。为模拟这一实际场景,在GEOM数据集的分子坐标中引入了受控噪声(图3a),以复现生成分子中常见的结构畸变。随后,利用该模型从这些受扰动的坐标中重建化学键。测试集上的评估结果表明,在存在噪声的条件下,模型仍能保持较强的性能,平均准确率、精确率、召回率和F1分数分别达到92.7%、93.6%、92.7%和92.7%(图3b)。
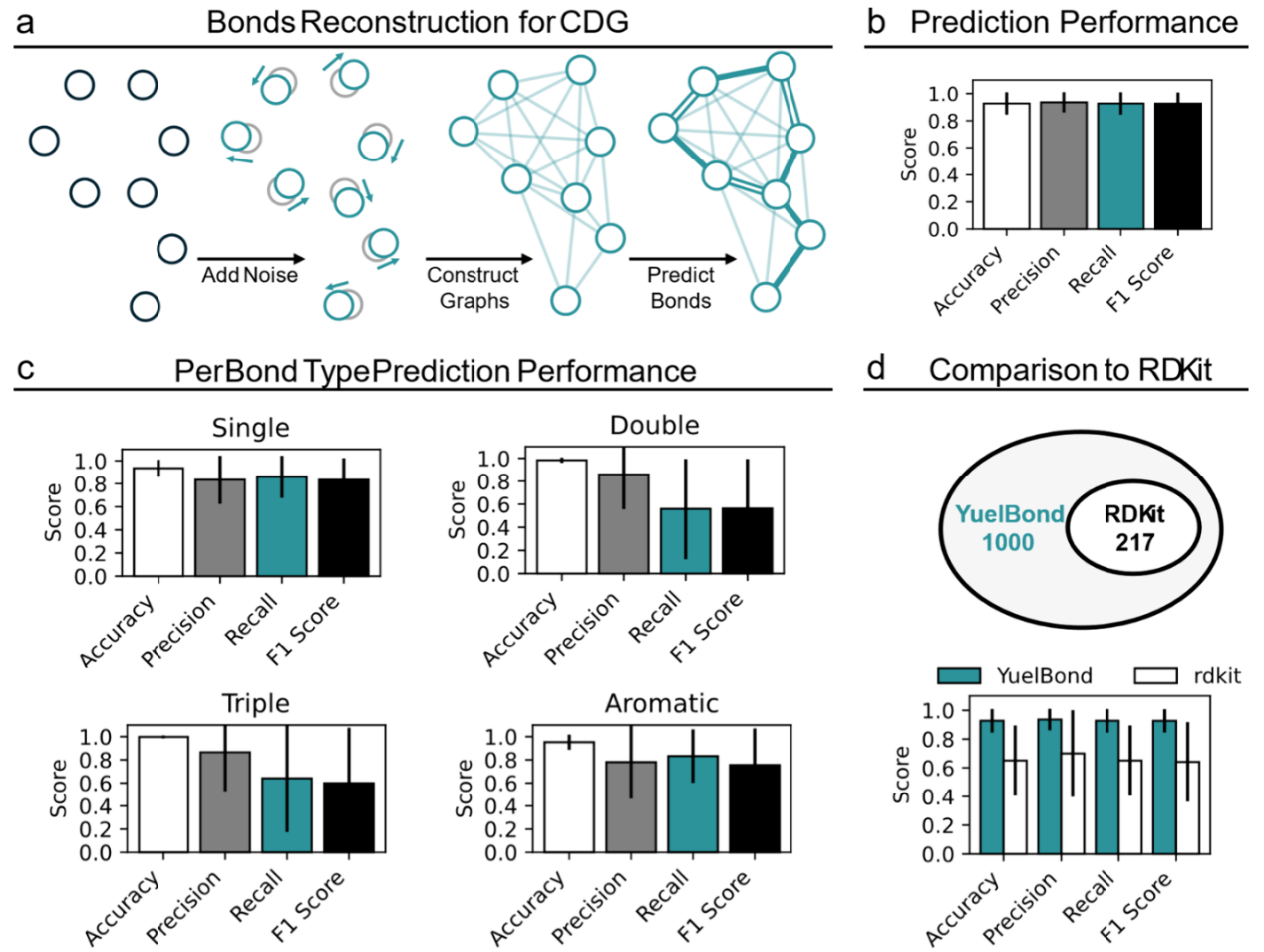
图3 YuelBond在CDG化学键重建任务上的性能评估
进一步分析了YuelBond在不同键类型上的预测性能(图3c)。结果表明,模型在所有键类型上均保持了较高的预测准确率,但精确率和召回率会随键的复杂程度而变化。单键由于出现频率高且局部几何特征明显,即使在受到扰动后仍然是预测最为可靠的键型。芳香键通常嵌入于具有显著拓扑特征的环结构中,因此尽管坐标存在噪声,仍能得到较好的预测。相比之下,双键和三键在结构畸变条件下面临更大的挑战,其预测高度依赖精确的键长和键角信息,而这些特征在结构噪声较大时变得不再可靠。因此,这两类键的召回率和F1分数较低,反映出假阴性数量的增加。
为对模型性能进行基准比较,将其与RDKit进行了对比(图3d)。在包含噪声坐标的1000个测试分子中,RDKit仅能成功处理其中的217个。即便在成功处理的217个分子中,RDKit也仅取得了65.3%的准确率、70.1%的精确率、65.3%的召回率和64.2%的F1分数,显著低于YuelBond。上述结果凸显了YuelBond在从不精确的三维结构中重建化学键级方面的鲁棒性,而在这一场景下,传统的基于规则的方法往往会失效。
二维生成化合物的化学键级重新分配
将YuelBond应用于仅基于原子连通性来推断化学键级(图4a)。在测试集上的评估结果表明,模型取得了80.1%的平均准确率、81.1%的精确率、80.1%的召回率以及78.3%的F1分数(图 4b)。
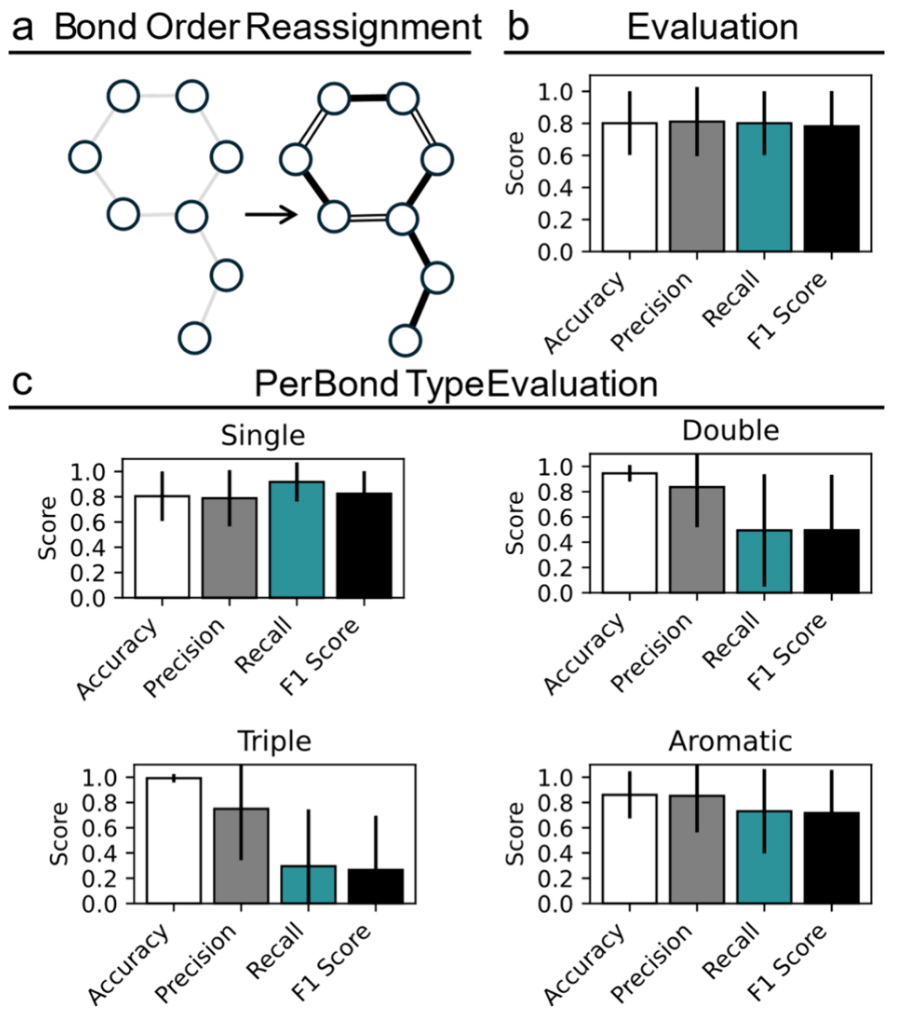
图4 YuelBond在化学键级重新分配任务中的性能评估
进一步考察了模型在不同键类型上的表现(图4c)。对于单键,YuelBond表现出较高的召回率和较好的F1分数,这反映了单键在分子图中具有高度一致性和普遍性。对于双键和三键,尽管准确率仍然较高,但由于其在数据集中相对少见,召回率较低。这是符合预期的,因为高阶键通常呈现出更为细微的模式,仅凭连通信息更难以区分。值得注意的是,三键的高准确率表明模型在避免假阳性方面具有很强能力,而较低的召回率则体现出一种偏向保守的预测策略,即更强调精确性而非过度预测。双键和三键相对于单键和芳香键更低的召回率,在很大程度上可归因于其在数据集中的稀有性。由于负样本(例如非三键)的数量远大于正样本,即便是较为保守的预测器也能获得较高的准确率。芳香键的预测表现较为均衡,表明其独特的连通模式在一定程度上被模型有效捕捉。
化学键级概率预测
尽管在化学键级重新分配场景中,双键和三键的整体预测准确率较低(图5a、b),但考察模型预测的置信度具有重要意义。以化合物(SMILES表示为O=CCC(=O)CCC#N)为例,计算了该分子中每一条化学键的预测概率(图5c)。例如,对于真实为三键的键1,模型将其误分类为单键,但仍为三键类别分配了27.3% 的概率,而单键和双键的概率分别为47.7% 和24.9%。这种对正确类别赋予相对较高概率的现象表明,即使最终预测结果未将其作为首选,模型仍保留了对正确键级的部分信心。相比之下,键2是一个正确预测的单键,其对应的预测概率高达90.3%,表明在歧义较小的上下文中模型具有很强的确定性。
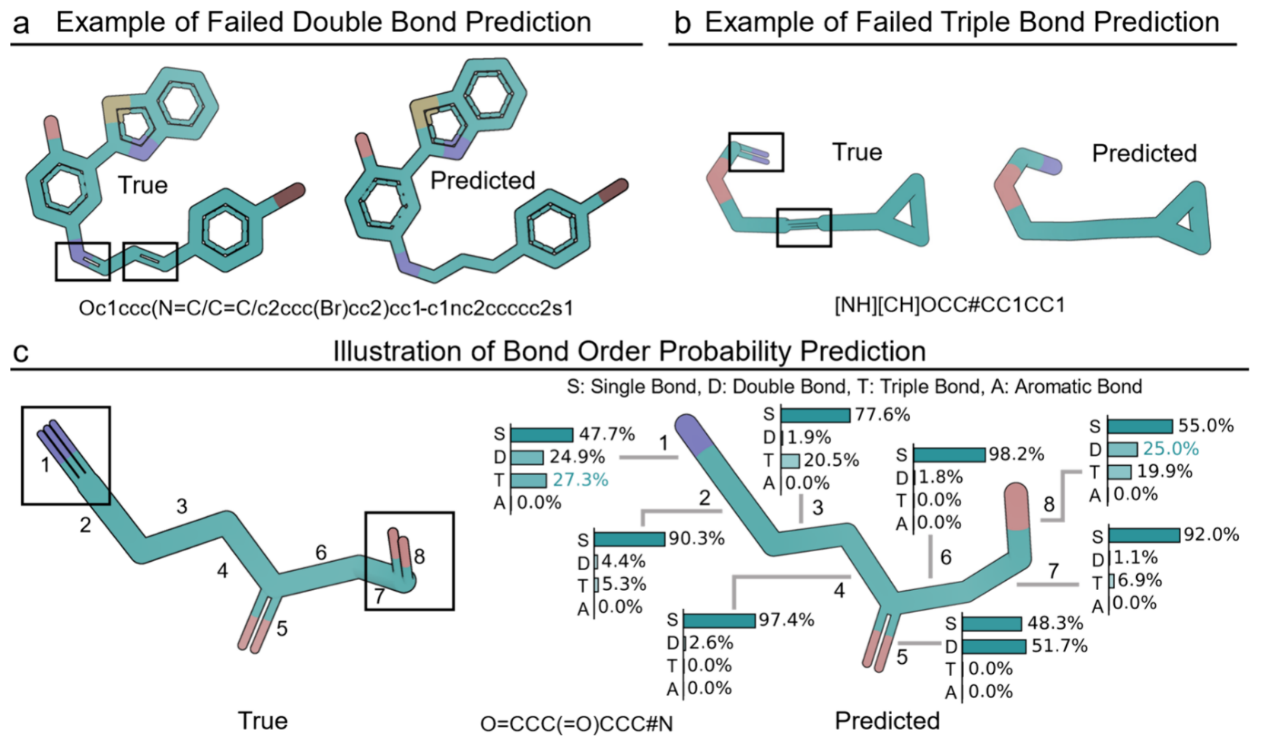
图5 化学键级概率预测
YuelBond优化对二维与三维生成分子的影响
使用DiGress(2D分子图生成模型)和DecompDiff(3D构象生成模型)生成了2000个原始分子结构,随后应用YuelBond对化学键级进行重新分配,并通过净化率和对数概率得分两项指标评估改进效果。
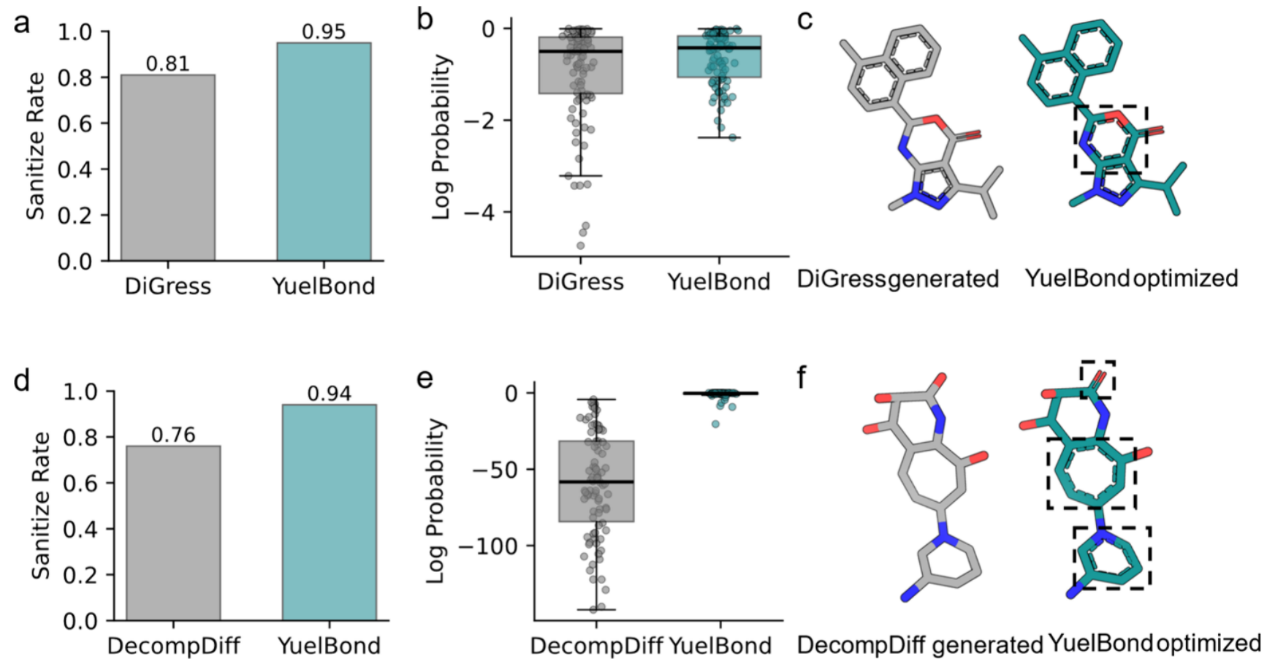
图6 YuelBond优化对2D和3D生成分子的影响
对于2D生成分子,DiGress输出的是具有明确原子连通性的分子图,但其原始输出的净化率仅为0.81,表明约20%的生成分子包含化学上无效的成键构型。在经过YuelBond优化后,净化率提升至0.95(图6a)。同时,优化后分子的对数概率得分在所有测试样本中均显著提高(图6b),表明YuelBond重新分配的键级显著增强了2D生成分子结构的化学合理性。对于3D生成分子,DecompDiff虽然能够生成原子坐标,但往往伴随几何结构畸变,从而导致化学键注释不可靠。这一问题直接体现在其较低的净化率0.76上(图6d),说明化学键生成仍是3D生成模型中的关键瓶颈。YuelBond的化学键重建将净化率提升至0.94,并同时提高了对数概率得分(图6e)。
Kekulé化数据集上的测试
在许多化学信息学应用中,尤其是基于图的处理流程中,通常会对芳香体系进行Kekulé化,即将芳香键转换为交替的单键和双键(图7a)。为评估模型在该设置下的鲁棒性,对GEOM数据集中的所有芳香键进行了Kekulé化处理,并基于该修改后的数据集重新训练了YuelBond,随后在三种预测场景中对其性能进行了评估。
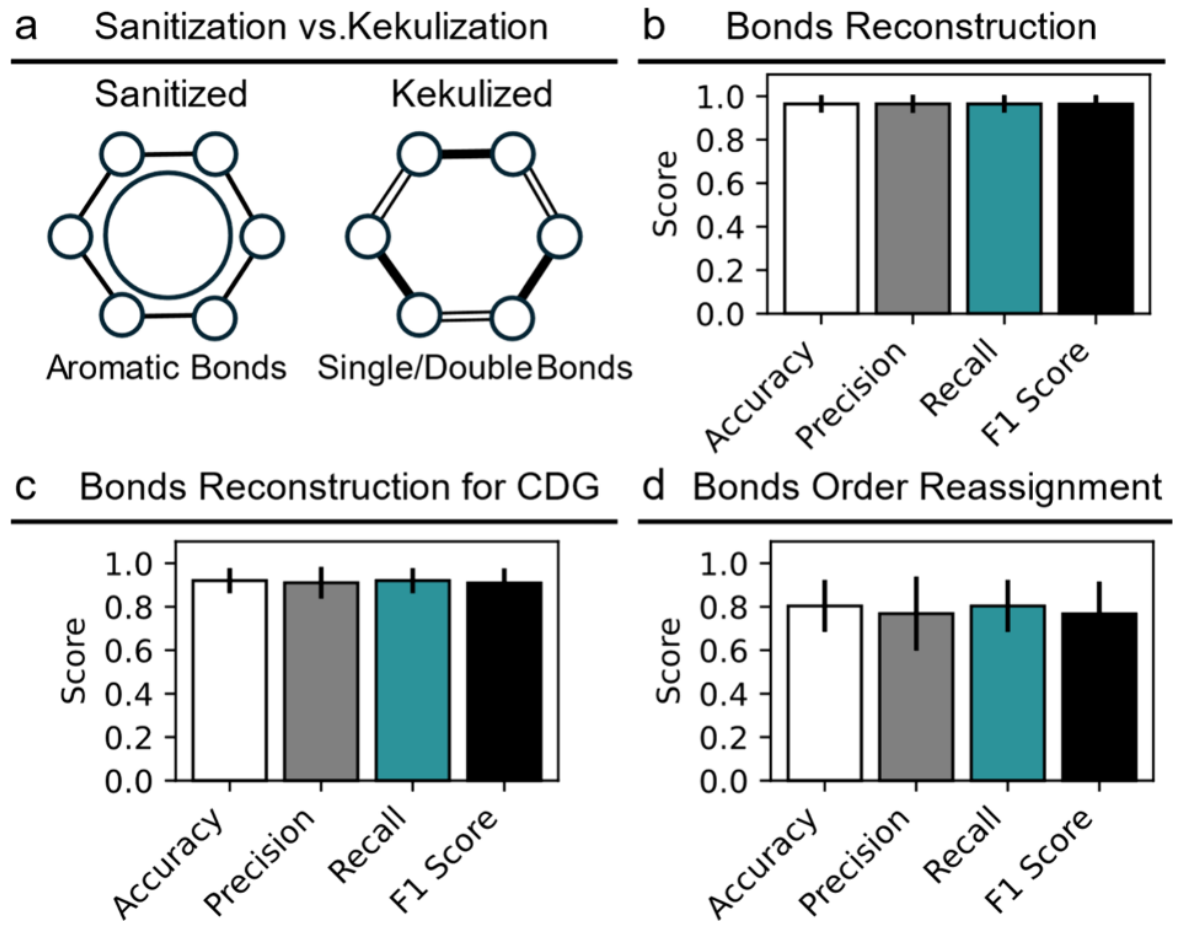
图7 YuelBond在Kekulé化数据集上的性能
实验结果由图7b-d所示,YuelBond的整体性能仍与其在非Kekulé化数据上训练的版本保持一致,表明该模型并不依赖于显式的芳香键标注,而能够在化学等价但表示方式不同的数据集之间实现良好的泛化。化学键级重新分配任务中F1分数的小幅下降,可能反映了在Kekulé化环体系中区分交替单键和双键所带来的额外歧义。总体而言,这些结果证实了YuelBond即使在Kekulé化数据条件下,仍能保持稳健而可靠的预测性能。
总结
YuelBond提供了一个统一的框架,能够在多种输入表示下学习并泛化化学成键规则。它在精确的三维场景中表现出色,能够容忍几何畸变,在二维分子图上也具有合理的适应能力,并同时提供具有可解释性的概率预测。YuelBond并非旨在完全取代基于规则的方法,而更应被视为一种互补工具,将机器学习的灵活性和上下文感知能力引入传统方法容易失效的应用场景中。
参考链接:https://doi.org/10.1021/acs.jcim.5c03052
文章改编转载自微信公众号:智药邦
原文链接:https://mp.weixin.qq.com/s/sh31E8-IN79ON0LKMuS3nA?scene=1&click_id=41 | 


























