药物–靶标相互作用(DTIs)是药物发挥治疗作用的基础,其准确预测有助于降低药物研发过程中实验筛选的成本和时间。现有的DTI预测方法通常侧重于分子拓扑结构的研究,这在一定程度上削弱了原子相对位置、键角等空间信息的表达能力,同时也未能有效融合分子信息与关联网络信息。
为解决上述问题,江南大学宁乔团队与大连海事大学李辉团队于2026年1月20日在《Journal of Chemical Information and Modeling》上发表文章,题为“Geometry-Enhanced Multiscale Joint Representation Learning for Drug-Target Interaction Prediction”。文章提出了一种新颖的几何增强多尺度联合表示学习方法GMJRL,用于药物–靶标相互作用预测。

GMJRL代码仓库:https://github.com/shhqiao/GMJRL/tree/v1.0
背景
药物研发是一个复杂且资源密集的过程,通常需要投入大量的时间、人力和资金,同时还伴随着较高的失败风险。准确预测药物–靶标相互作用能够有效缩小候选药物分子的筛选范围,从而加速药物研发进程。传统依赖生物实验的候选药物发现方法不仅耗时,而且成本高昂。因此,近年来计算方法受到了越来越多的关注。
分子对接是预测药物–靶标相互作用的重要方法之一,但其受限于许多靶标蛋白缺乏已知的三维结构。传统机器学习方法主要基于核方法、聚类、矩阵分解以及网络模型。然而,随着生物活性数据的不断增长与药物–靶标相关的数据分布变得愈加复杂,传统机器学习方法难以有效刻画这种复杂性,而深度学习在处理复杂数据分布方面具有显著优势。基于深度学习的方法通常从序列、结构或网络中提取药物和靶标的表示,但并未深入挖掘药物和靶标的几何结构信息。尽管已有研究尝试利用几何信息增强药物和靶标表示,但仍未能同时整合靶标的全局结构关系与局部空间坐标信息。此外,这类方法往往存在单一视角表示不足的问题。与之不同的是,本文从微观尺度和宏观尺度同时提取药物和靶标的属性(图1),并构建了一种用于融合的联合表示方法。
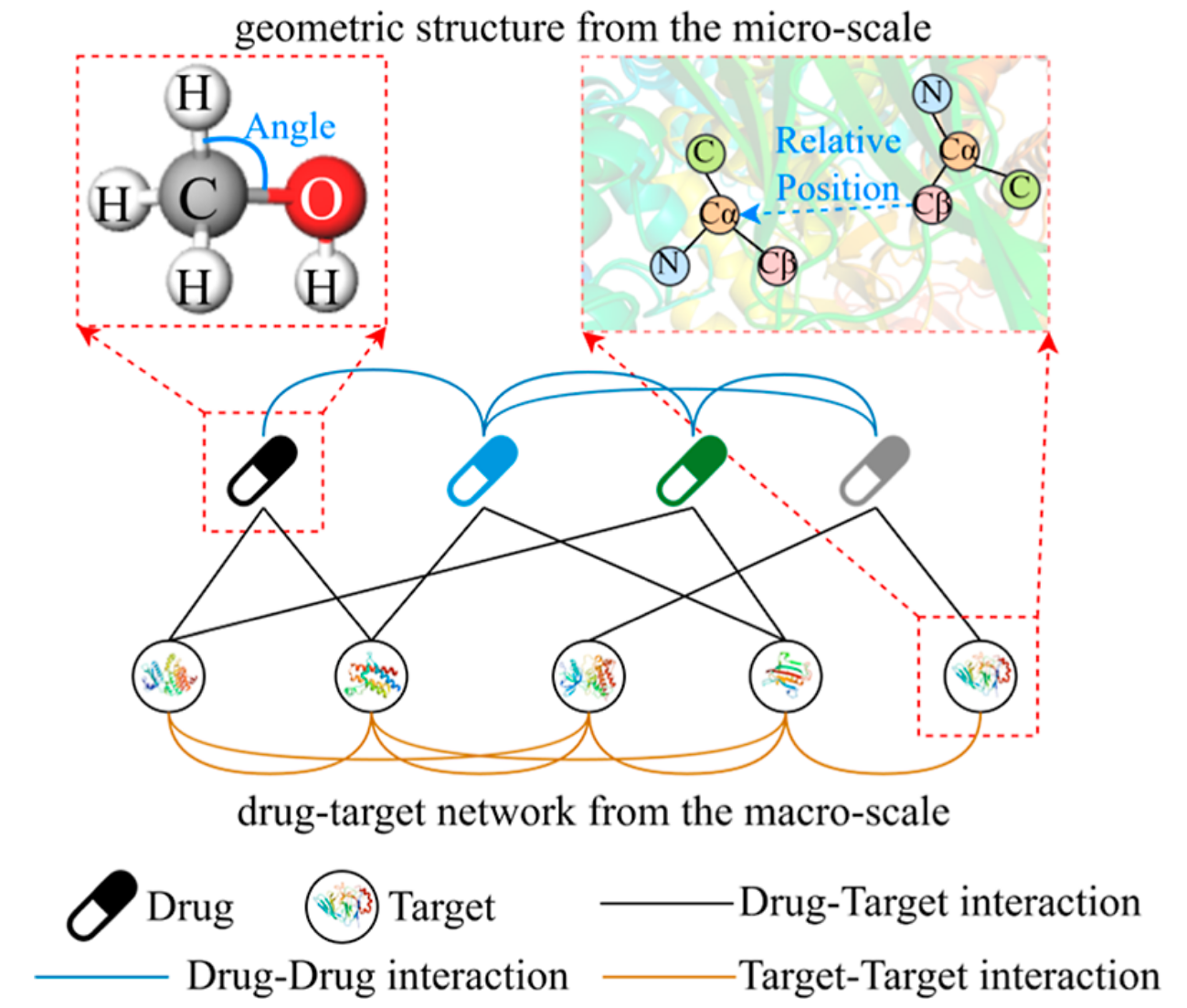
图1 药物和靶标在微观尺度与宏观尺度下的属性
方法
GMJRL整体框架如图2所示。具体来说,GMJRL首先从微观视角学习药物和靶标的几何结构表示:对于药物,引入化学键角信息(图2a);对于靶标,将基于GCN的局部结构表示、自注意力机制的全局相互作用建模以及残基层面的三维坐标编码到同一表示学习框架中(图2c)。其次,GMJRL构建异构药物–靶标网络,并从宏观视角提取网络表示(图2b)。随后,提出一种基于自注意力机制的联合表示学习方法,在融合结构表示与网络表示的过程中,同时考虑尺度内相关性与尺度间相关性,从而实现多尺度表示的有效整合(图2d)。最后,GMJRL提出了一种负采样算法(图2e),通过提供可靠的负样本来提升模型的DTI预测能力(图2f)。
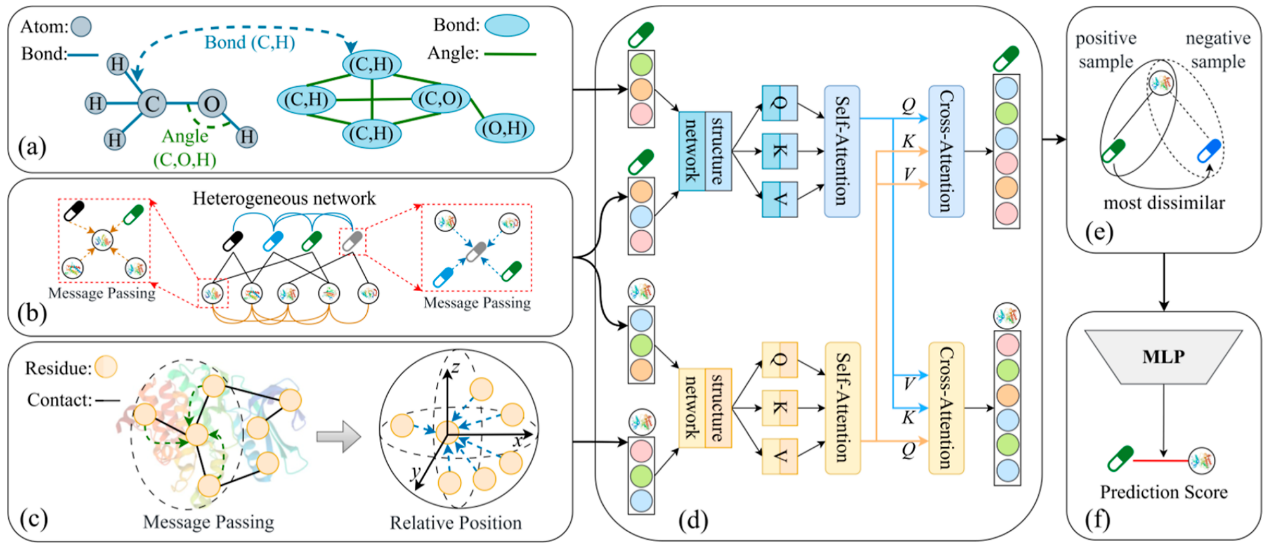
图2 GMJRL框架图
结果
数据集
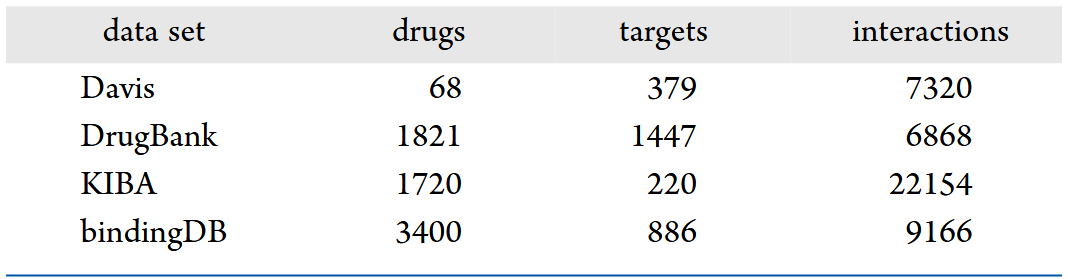
在四个基准数据集上对GMJRL进行了全面评估,数据集分别为DrugBank、Davis激酶抑制剂数据集、激酶抑制剂生物活性(KIBA)数据集以及BindingDB数据集。表1展示了上述数据集的基本信息。
表1 本研究所使用数据集的总体概况
与基线方法比较
在四个数据集上将GMJRL与多种基线模型进行了对比,具体的指标数值列于表2–5中,整体性能对比结果如图3所示。
表2 GMJRL与基线模型在Davis数据集上的性能对比
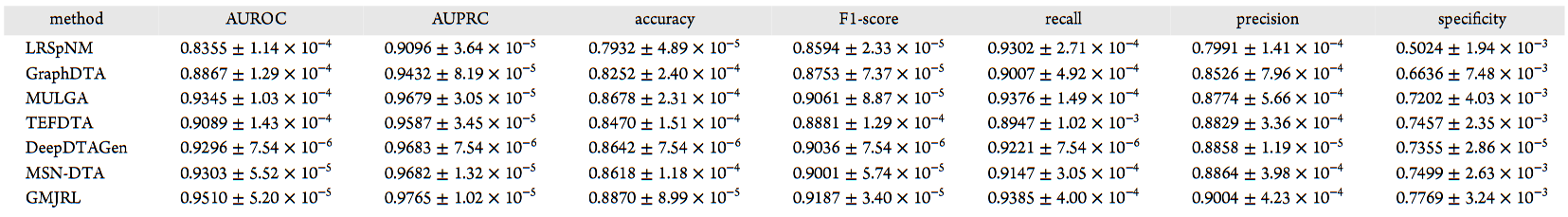
表3 GMJRL与基线模型在KIBA数据集上的性能对比
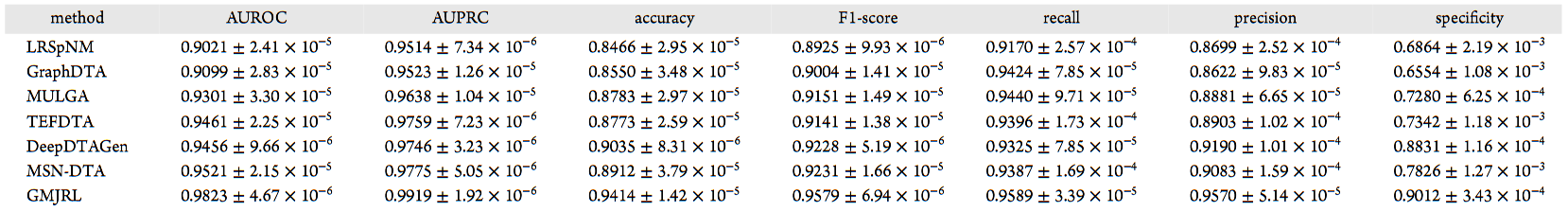
表4 GMJRL与基线模型在BindingDB数据集上的性能对比
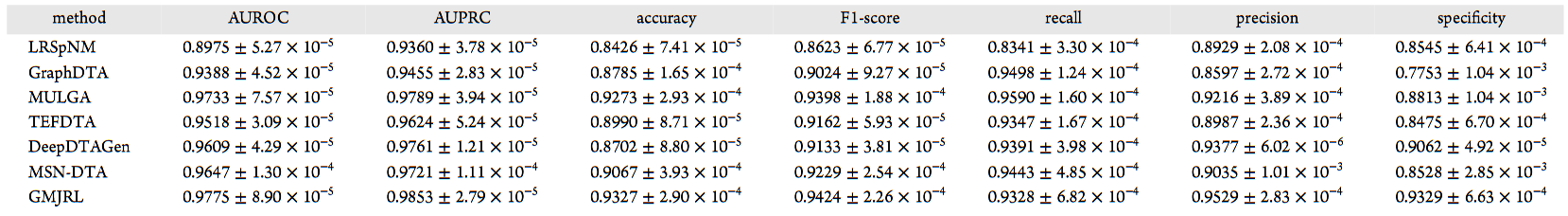
表5 GMJRL与基线模型在DrugBank数据集上的性能对比
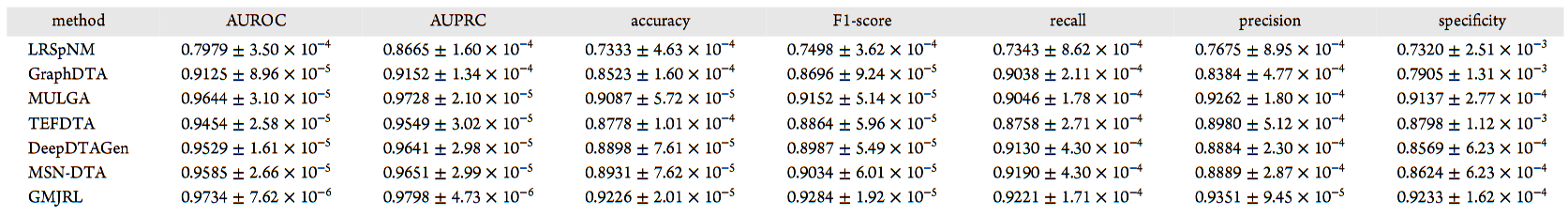
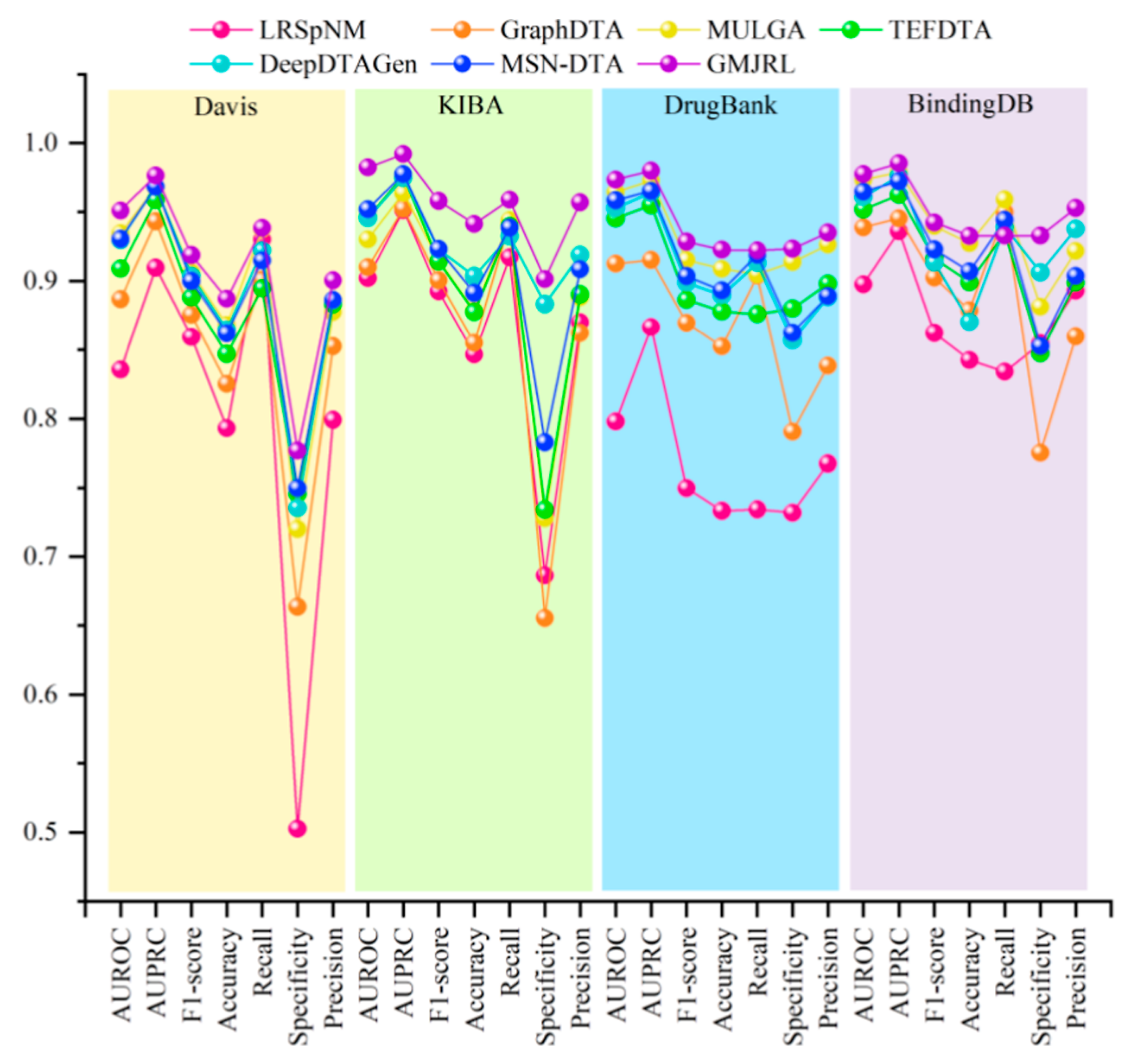
图3 GMJRL与基线方法在四个数据集上的性能对比
实验结果表明,GMJRL在Davis、KIBA和DrugBank数据集上的所有评价指标均取得了最佳性能;在BindingDB数据集上,除召回率(Recall)外,其余指标均优于对比方法。尽管其他模型也取得了较好的预测效果,但它们未能进一步挖掘药物和靶标的几何结构信息,且由于依赖单一类型信息,仍存在表示能力不足的问题。相比之下,GMJRL不仅深入刻画了药物和靶标的几何结构信息,还通过多尺度联合表示学习灵活融合结构表示与网络表示,使模型能够从多个视角捕获药物–靶标特征,从而获得更加全面的表示。实验结果充分表明,GMJRL在药物–靶标相互作用预测任务中展现出卓越的性能。
消融实验
本文开展了模块的消融实验。具体而言,GMJRL-w/o GSR移除几何结构表示模块;GMJRL-w/o INR移除相互作用网络表示模块;GMJRL-w/o IIE移除相互作用信息提取模块。
如图4A所示,完整的GMJRL模型在性能上优于所有变体模型。几何结构表示有助于模型理解药物–靶标的结构特性,提供关键的分子层级信息;相互作用网络表示能够捕获药物与多个靶标之间的关系,有助于识别潜在相互作用;相互作用信息提取模块则进一步建模药物与靶标之间的交互关系,增强模型对复杂关联的理解。实验结果表明,各模块均在模型性能提升中发挥了重要作用。
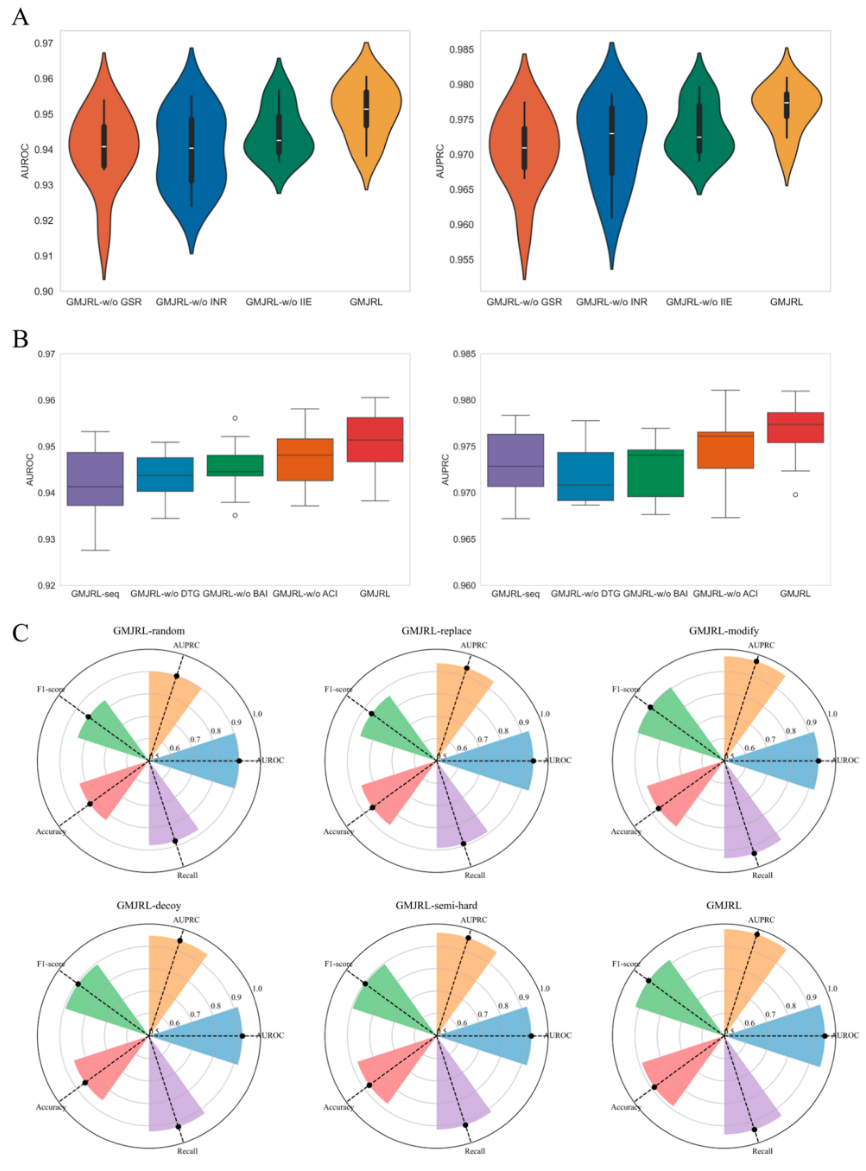
图4 GMJRL的消融实验、几何信息性能验证以及负采样策略验证实验结果
几何信息性能验证
为评估引入几何信息所带来的性能提升,进一步设计并测试了以下GMJRL变体模型。具体而言,GMJRL-seq将药物和靶标的几何结构特征提取替换为序列特征提取;GMJRL-w/o DTG同时移除药物和靶标的几何信息;GMJRL-w/o BAI仅移除药物中的键角信息;GMJRL-w/o ACI仅移除靶标中的原子坐标信息。
如图4B所示,移除任意一种几何信息都会导致模型性能下降。去除键角信息会简化药物的空间构象,使其难以准确反映真实的三维形态;去除靶标的坐标信息则会使模型无法精确刻画靶标的真实结构和空间分布,从而降低药物–靶标相互作用预测的准确性。即使在不使用三维结构信息的情况下,模型仍能保持较好的性能,但引入三维几何信息后可进一步提升预测效果。此外,GMJRL的性能优于GMJRL-seq,这是因为结构图能够编码更为丰富的三维空间信息和立体相互作用模式,从而更准确地表征药物–靶标的结合方式和作用机制,最终提升预测精度。
负采样策略性能验证
为评估本文提出的负采样策略的有效性,引入并测试多种GMJRL变体开展了一系列对比实验。具体而言,GMJRL-random从未标注的DTI中随机选取负样本;GMJRL-replace采用Wu等人提出的负采样策略,该方法首先排除与给定药物相似药物所关联的靶标,然后从剩余靶标中随机选择一个作为该药物的负样本;GMJRL-modify对负采样算法进行改进,其核心思想是:若某一靶标能够与给定药物结合,则与该靶标最不相似的靶标不太可能与同一药物结合;GMJRL-decoy针对每个正样本药物,选择属性相似但结构不同的药物来构建负样本;GMJRL-semihard针对当前药物,首先选取与其结构相似的药物,并收集这些相似药物的相互作用靶标作为候选负靶标,随后从中选择与当前药物的距离大于正样本距离、但仍会产生损失的样本作为负样本。
从图4C可以看出,本文提出的负采样策略取得了最佳性能。相比之下,本文提出的负采样策略更符合生物学实际,有效降低了无关靶标的干扰。此外,如图5所示,采用本文负采样策略训练得到的模型,其表示空间具有更加清晰的决策边界和更强的判别能力。综上所述,实验结果表明,本文提出的负采样策略是一种更优的采样方案,能够显著提升预测准确性。
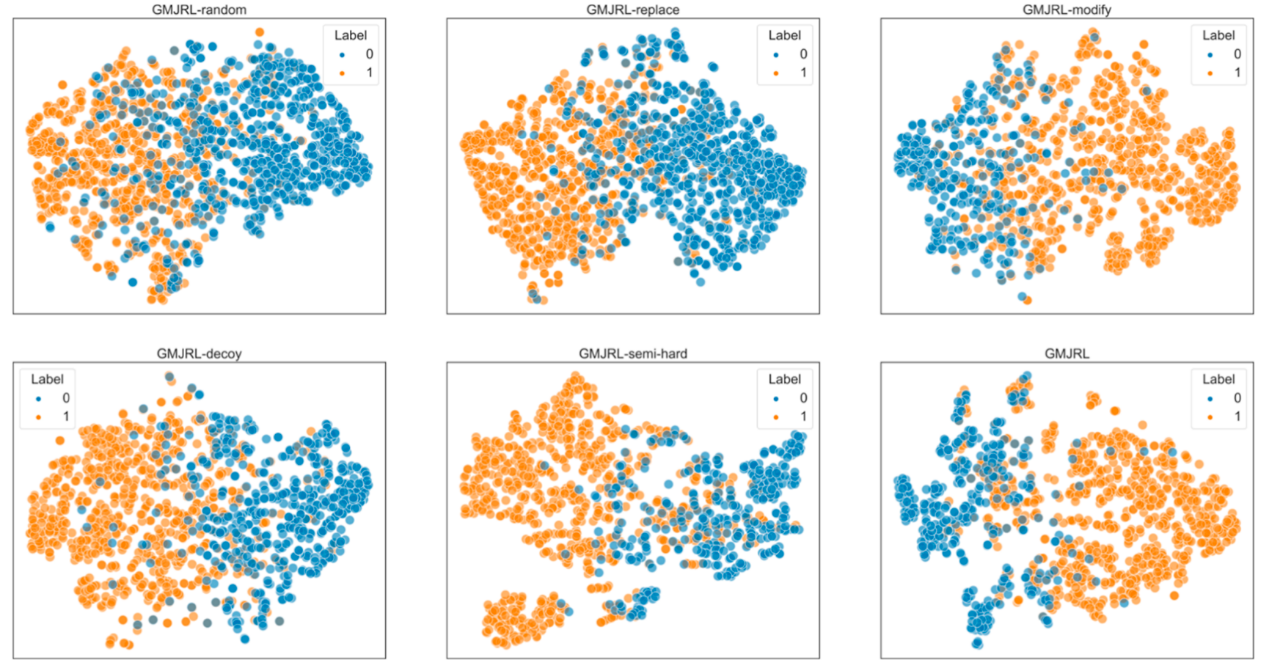
图5 四种负采样策略下特征空间分布的t-SNE可视化结果
多尺度联合表示性能验证
为评估多尺度联合表示学习的效果,本文设计并测试了以下四种GMJRL变体模型。具体而言,GMJRL-concate直接拼接两种尺度的表示;GMJRL-addition对两种尺度的表示进行加和;GMJRL-λ₁λ₂引入两个可学习参数对不同尺度表示进行融合;GMJRL-λ(1−λ)引入一个可学习参数对不同尺度表示进行融合。
根据图4A的结果可以观察到,任何多尺度联合表示方法的性能均优于仅使用单一尺度表示的模型。如图6A所示,本文采用的多尺度联合表示方法取得了最佳性能。这是由于拼接、求和以及引入可学习参数等融合方式虽然简单有效,但无法动态调整不同尺度表示之间的权重和关系;而自注意力机制通过建模尺度内与尺度间的相关性,实现了更加自适应且表达能力更强的特征融合,因此在多尺度联合表示学习任务中表现更优。
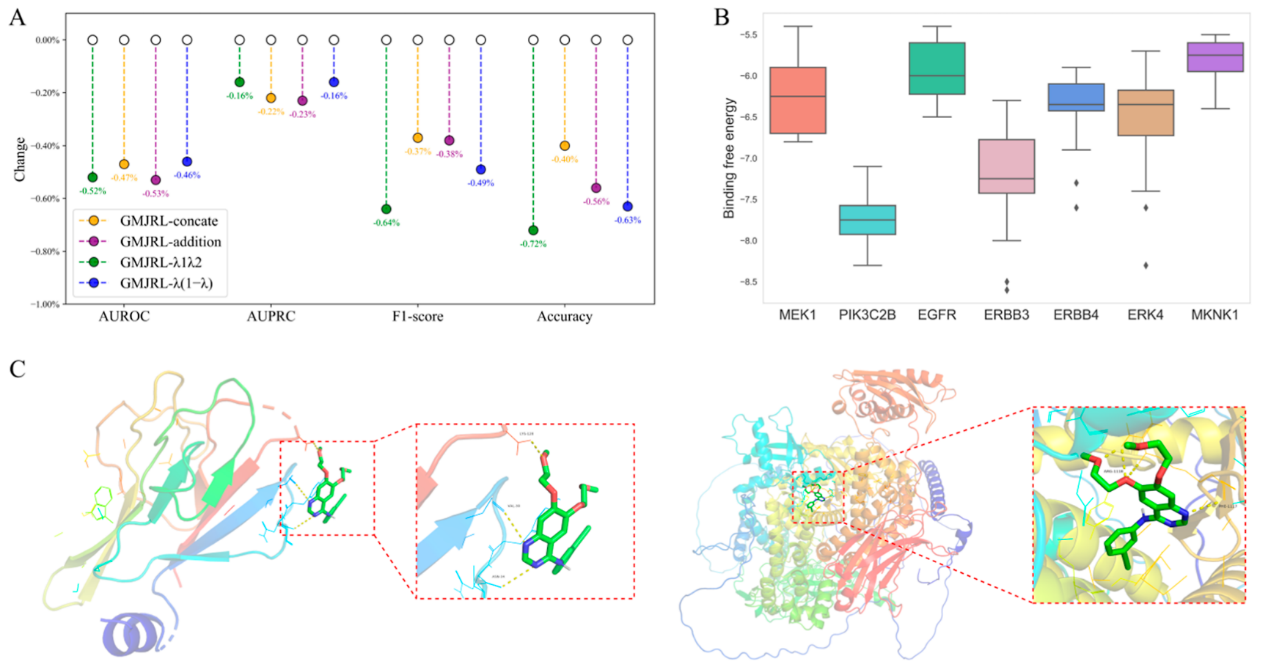
图6 GMJRL的多尺度联合表示性能验证实验
案例研究
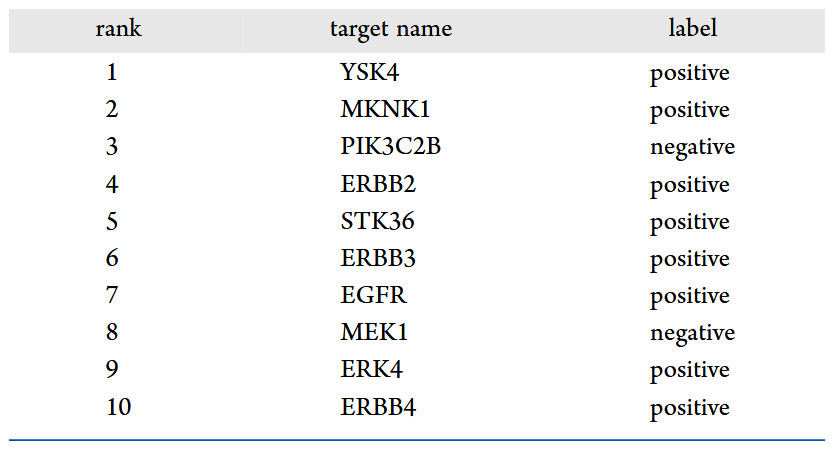
以埃罗替尼(Erlotinib)为研究对象,该药物是一种酪氨酸激酶抑制剂,常用于治疗晚期或转移性胰腺癌和非小细胞肺癌。表6列出了GMJRL预测的埃罗替尼的前10个潜在靶标,其中有8个为已知关联靶标。对于其余两个潜在靶标MEK1和PIK3C2B,进一步进行了分子对接分析。图6C所示的对接结果表明,埃罗替尼能够与这两个靶标的氨基酸残基形成分子间相互作用。此外,比较了埃罗替尼与MEK1、PIK3C2B以及5个已知关联靶标之间的对接评分(图6B)。结果显示,埃罗替尼与MEK1的对接评分与已知靶标相当,而其与PIK3C2B的对接评分则明显低于已知靶标。MEK1和PIK3C2B均位于EGFR信号通路的下游,作为EGFR抑制剂,埃罗替尼可能通过抑制EGFR的激活,间接抑制MEK1和PIK3C2B信号通路的活性,从而影响肿瘤细胞的增殖、生存和侵袭能力。
表6 预测的Erlotinib候选靶标
几何信息在区分高度相似药物中的作用
为评估几何信息在GMJRL中区分相似药物方面的贡献,本文选取了Canertinib和Gefitinib作为测试案例,这两种药物的MACCS指纹相似度高达84%(图7a)。分别使用GMJRL及其移除几何信息的消融模型GMJRL-w/o DTG对两种药物的潜在靶标进行预测。
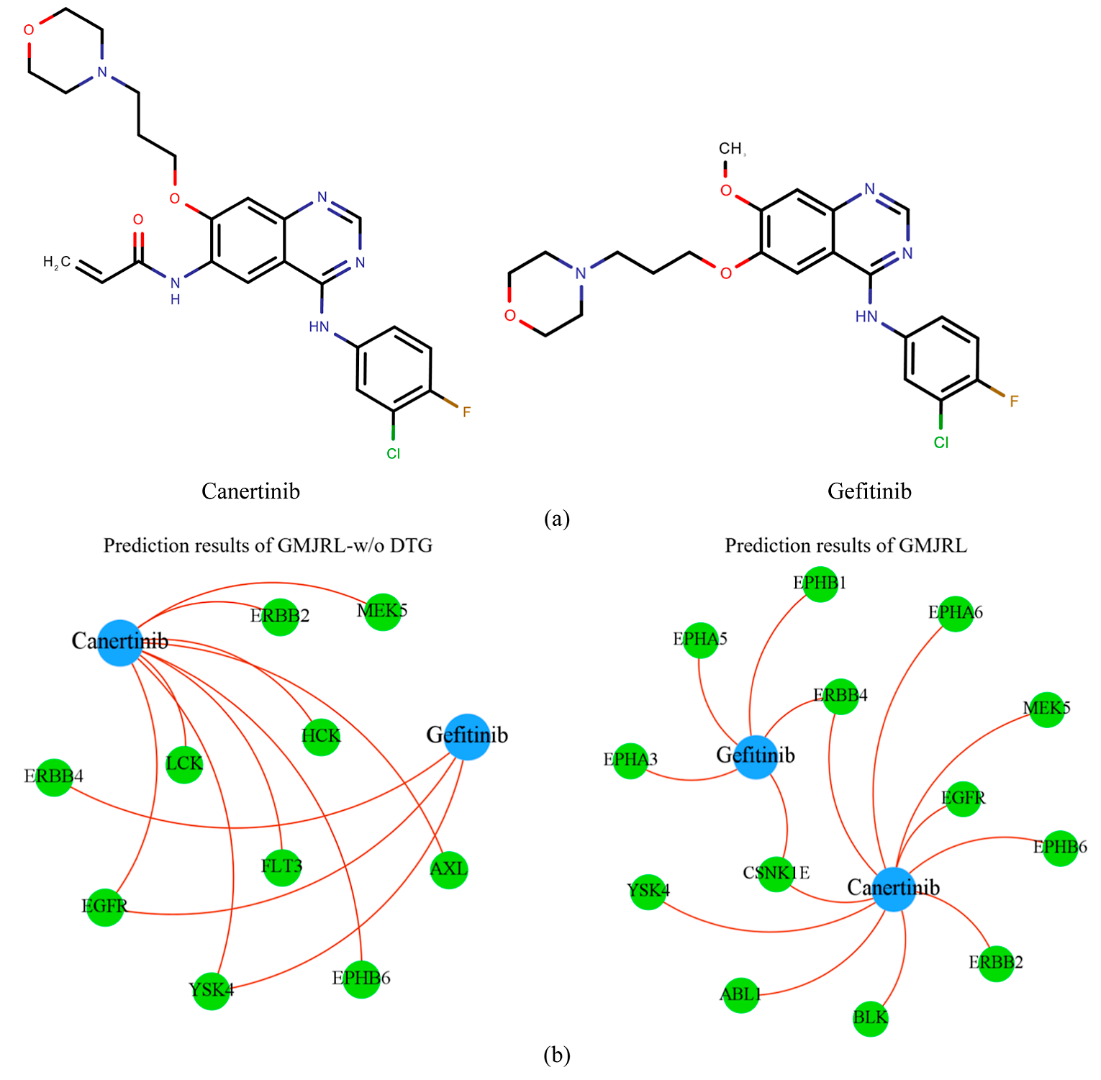
图7 Canertinib与Gefitinib的分子结构以及两种模型的预测结果
图7b展示了两种模型在各自预测的前10个靶标中识别出的真实已知靶标数量。结果表明,引入几何信息显著提升了模型区分高度相似药物的能力。由于药物功能依赖于其三维结构与靶标之间的空间互补性,表面结构相似的药物在几何构象、结合构型及相互作用模式上往往存在本质差异。通过整合几何信息,GMJRL能够更准确地捕捉这些细微的结构差别,从而提升对结构相近化合物靶标识别的精度。因此,几何信息是增强模型区分相似药物能力的关键因素。
总结
本文提出了一种基于几何增强多尺度联合表示学习的全新方法GMJRL。该方法利用几何神经网络引入药物中的化学键角信息以及靶标中的原子坐标信息,从而深入挖掘药物与靶标的三维几何结构特征。在此基础上,GMJRL基于自注意力机制设计了多尺度联合表示学习框架,对结构表示与网络表示进行融合,获得更加鲁棒的药物–靶标联合表示。此外,本文还提出了一种能够有效筛选可靠负样本的负采样算法。实验结果表明,GMJRL在药物–靶标相互作用预测任务中表现优异,且三维几何信息能够显著提升模型的预测性能。未来的研究将从以下几个方面进一步提升GMJRL的DTI识别能力:一是探索更丰富的几何信息,以进一步增强药物和靶标的结构表示;二是设计更加高效且具有普适性的多尺度联合表示学习方法。
参考链接:https://doi.org/10.1021/acs.jcim.5c02347
--------- End ---------
文章改编转载自微信公众号:智药邦
原文链接:https://mp.weixin.qq.com/s/ts0Pwnuxce_yEOSr6qlXmA | 


























